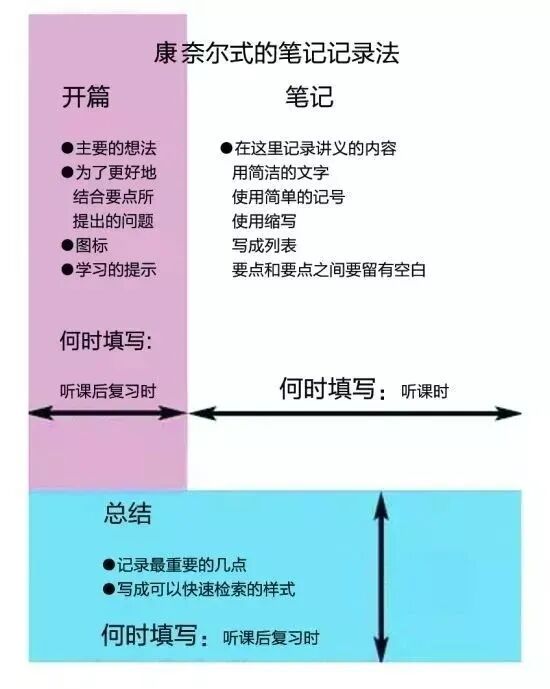
一、导师简介

Prof. Kei Hang Katie CHAN(陈纪行)现任香港城市大学电机工程学系与生物医学系双聘副教授。其学术训练呈现清晰的跨学科轨迹:于香港大学取得信息工程学士学位后,转向公共卫生领域,在南加州大学获得流行病学与生物统计学硕士,继而以Burroughs Wellcome Fund代谢性疾病奖学金资助完成加州大学洛杉矶分校流行病学博士训练,专攻2型糖尿病与心血管疾病的遗传结构。
博士后期阶段,Prof. CHAN在UCLA与布朗大学开展博士后研究,系统识别出2型糖尿病与心血管疾病间的共享分子通路与基因网络。2016年返回香港后,先后任职于香港中文大学糖尿病与肥胖研究所、布朗大学全球心脏代谢健康研究中心,2018年正式加入香港城市大学建立独立课题组。
这种跨越工程学、公共卫生、流行病学的训练背景,塑造了其研究特色的两大支点:其一,将复杂疾病视为多层级分子网络扰动的结果而非单因素病理过程;其二,强调计算工具必须服务于临床可解释性。其课题组研究兴趣集中于遗传与分子流行病学、系统生物学、计算生物学及生物信息学,核心方法论为整合多组学变异、生物标志物及环境数据,构建疾病复杂网络模型。
二、近期文章和项目解析

Prof. CHAN在2024-2025年间发表60项研究成果,涵盖期刊论文、会议报告与专利。以下针对代表性工作进行技术解析:
1. 自身免疫疾病共享遗传架构的多组学验证(Functional & Integrative Genomics, 2025)
该研究针对类风湿关节炎(RA)、多发性硬化症(MS)与1型糖尿病(T1D)三种自身免疫疾病,整合全基因组关联研究(GWAS)、基因表达综合数据库(GEO)、分子特征数据库(MSigDB)及单细胞RNA测序(scRNA-seq)数据。研究流程包含三个阶段:首先,通过差异基因表达分析与LASSO回归从全血组织批量RNA-seq数据中筛选共享因子,锁定活性氧物种(ROS)通路相关基因_ROMO1_;其次,利用多样本GEO数据集验证_ROMO1_与免疫模式的跨组织一致性;最后,采用双样本孟德尔随机化(MR)分析确立_ROMO1_对单核细胞表位的调控作用。
此项工作的技术贡献在于:通过单基因ROC曲线分析验证_ROMO1_作为疾病生物标志物的判别效能,AUC值在训练集与验证集中均保持稳健;单细胞层面解析显示_ROMO1_在单核细胞中表达显著偏离健康对照,且在三种疾病中呈现一致的免疫浸润模式。该研究将ROS通路的代谢失调与自身免疫发病机制直接关联,为跨疾病干预靶点提供了遗传学证据。
2. 宫颈癌生存预测模型的算法比较(Scientific Reports, 2025)
基于SEER数据库构建五种生存预测模型,比较梯度提升、随机森林等传统机器学习方法在五年病因特异性生存预测中的表现。研究采用107,473例宫颈癌患者数据,涵盖2000-2019年诊断案例,纳入年龄、分期、组织学类型、治疗方式等37个临床变量。
模型评估显示,集成学习方法在判别能力(C-index)与校准度方面均优于单一算法。其中,XGBoost模型在验证集中AUC达0.787,C-index为0.773,显著高于Cox比例风险模型的0.721。特征重要性排序表明,手术方式、淋巴结转移数量与肿瘤分级为前三位预测因子。该研究的临床价值在于证明:即便在数据质量参差不齐的真实世界登记数据库中,适当选择的机器学习算法仍能提取稳健的风险分层信号。
3. 流感病毒交叉免疫的深度学习预测(Journal of Automation and Intelligence, 2025)
针对H3N2亚型抗原漂移导致的交叉免疫保护难题,开发预训练深度学习模型DPCIPI。该模型采用双向长短期记忆网络(Bi-LSTM)编码病毒血凝素蛋白序列,结合历史流行病学数据与抗原表位变异信息,预测新毒株与既往毒株间的交叉免疫水平。
技术架构上,DPCIPI引入注意力机制捕捉关键抗原位点,模型在2010-2023年季节性流感数据上训练,交叉验证显示预测相关系数达0.84。与传统序列比对方法相比,深度学习模型有效捕捉了表位共变异等非线性模式,为疫苗株选择提供计算辅助决策工具。
4. 认知障碍营养干预的机制验证(Clinical Nutrition ESPEN, 2025)
纳入52项随机对照试验共8452名参与者,结合网络荟萃分析(NMA)与孟德尔随机化(MR)评估营养干预对轻度认知障碍(MCI)与阿尔茨海默病(AD)的效果。NMA结果显示多成分复合干预效果最优(标准化均值差SMD=2.03, 95%CrI:0.97-3.09),MR分析进一步揭示此类干预通过降低C反应蛋白(CRP)水平发挥抗炎作用(OR=0.96, 95%CI:0.93-0.99)。
该研究的创新性体现于方法学整合:NMA处理直接比较证据,MR则利用遗传变异作为工具变量推断因果方向,两者结合超越了传统meta分析只能建立关联的局限。从GWAS数据提取的9种营养素遗传预测得分与12项脑衰老生物标志物的关联分析,为"饮食-炎症-认知"通路提供了遗传学支持。
5. 自闭症谱系障碍药物重定位研究(European Journal of Pharmacology, 2025)
构建血液与脑组织的蛋白-蛋白相互作用网络(PPI),识别自闭症谱系障碍(ASD)关键驱动基因。研究整合GTEx数据库脑组织表达谱与ASD患者外周血转录组数据,通过WGCNA算法构建共表达模块,筛选模块hub基因。
基因集富集分析显示,血液与脑组织共享的驱动基因集中于突触可塑性、神经炎症与线粒体功能通路。基于这些基因的药物-基因互作网络分析,筛选出5种已获批药物(包括二甲双胍、雷帕霉素等)具有重定位潜力。该研究的转化意义在于:利用易获取的外周血样本推断中枢病理机制,降低药物开发成本。
6. 技术专利布局分析
Prof. CHAN团队同期申请三项专利,覆盖疾病诊断、药物开发与因果推断工具。其中,申请号63/864,343的专利构建胰腺癌机器学习管道,整合TCGA与GEO数据实现基因预测与药物重定位一体化;申请号19/270,740的专利开发识别因果关系算法,可能应用于观察性研究的混杂控制。
三、未来研究预测
基于现有研究轨迹与技术积累,未来工作可能沿以下脉络展开:
其一,多组学整合深度将进一步增强。当前研究多基于转录组与遗传数据,未来可能融入蛋白质组、代谢组及微生物组,构建真正"多层级"疾病网络。特别是单细胞空间转录组技术的应用,可在组织微环境层面解析细胞间通讯网络,为复杂疾病提供更高分辨率的机制图谱。
其二,预测模型向动态化与个体化发展。现有生存预测多为静态基线模型,未来可结合电子健康记录实时数据,开发时变风险预测系统。个体化层面,通过整合个人基因组数据与纵向监测指标,构建"数字孪生"疾病模型,实现精准预防。
其三,从疾病相关性迈向因果干预。孟德尔随机化方法的应用表明课题组重视因果推断,未来可能开展孟德尔随机化引导下的临床试验设计(MR RCT),利用遗传证据优化干预靶点选择,提升研究转化效率。
其四,计算工具临床部署加速。多项专利申请显示团队注重技术转化,未来可能推出可临床应用的决策支持系统,如流感疫苗选型工具、癌症辅助治疗推荐系统等,实现从出版物到临床产品的闭环。
其五,跨疾病机制研究持续深化。ROMO1基因在三种自身免疫疾病中的共享作用提示,课题组可能继续挖掘代谢-免疫交互界面在其他疾病簇(如心血管-代谢-肾病综合征)中的核心调控节点,推动"疾病组学"(diseasome)层面的网络医学实践。