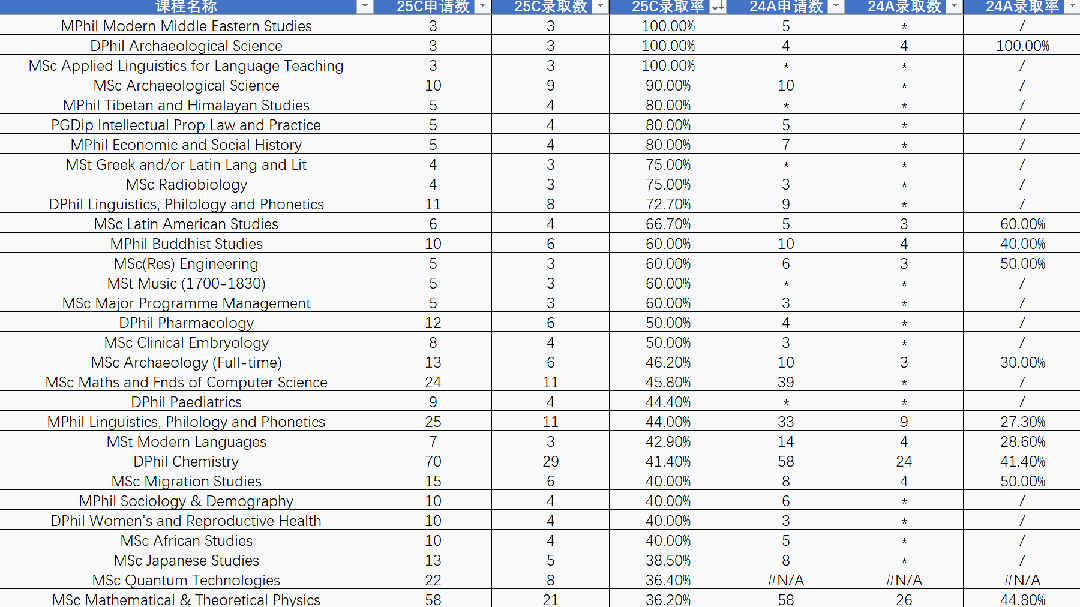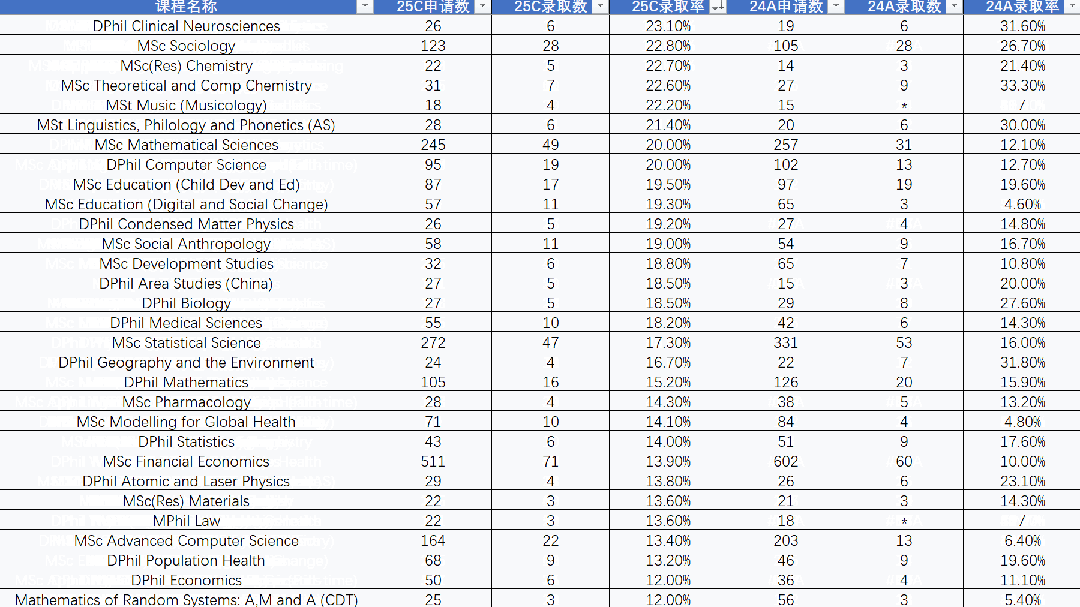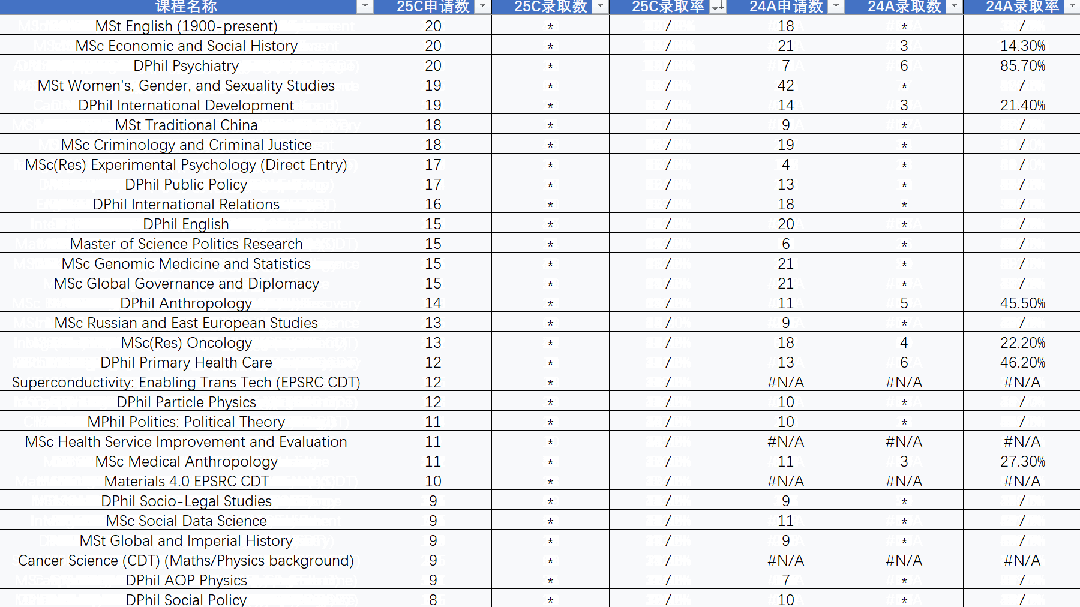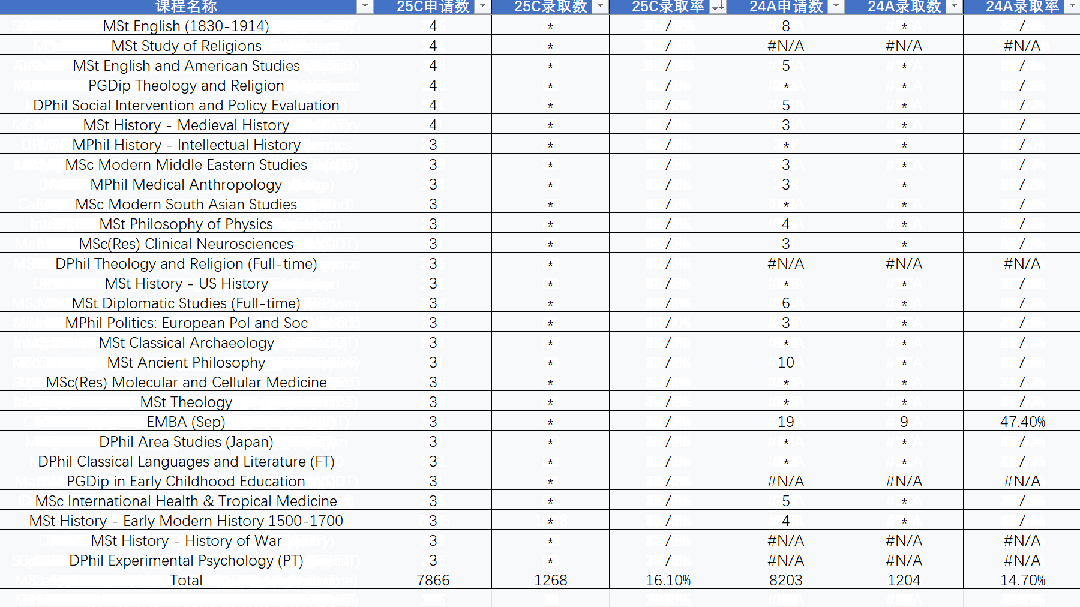
2023 iGEMiGEM课题分享
2016 Imperial_College ecolibrium

Achievements:Grand Prize Winner Undergraduate,
Best Foundational Advance Project,
Best Wiki,
Best Poster,
Best Education&Public Enga-gement,
Best New Basic Part.
Part.01项目背景Background
自然界中,不同种微生物生活在同一个生态环境中,单独隔绝的微生物种群是不存在的。不同种微生物之间也存在信息交流与能量利用,这对微生物群的生长调控至关重要,同时也有利于最大化利用环境资源。
而实验室在一体系中定量培养多种微生物是很困难的。因为不同微生物最佳生长条件有差异,在共培养体系中微生物培养条件难以确定,且更加困难的是做到精确调控不同种微生物群之间的期望比例。
Imperial_College想针对共培养技术所遇到问题,设计一个可精确调控菌群比例的共培养系统。
Part.02课题设计design
团队设计了三个模块以构建可调控的共培养系统,分别是通信模块、比较模块、生长调节模块。
除了基因线路,Imperial_College还设计了A.L.I.C.E.软件,帮助研究者确定共培养条件。
1.通信模块
通信模块利用两个正交的群体感应系统,使我们的大肠杆菌种群能够检测自己的种群密度以及其他种群的种群密度。
为了允许在电路中使用不同的群体感应系统,我们选择使用四种可能的系统:Las,Rhl,Lux和Cin,分别被30-C12AHL、C4AHL、30-C6AHL、30-C14AHL群体感应调节因子所激活诱导。
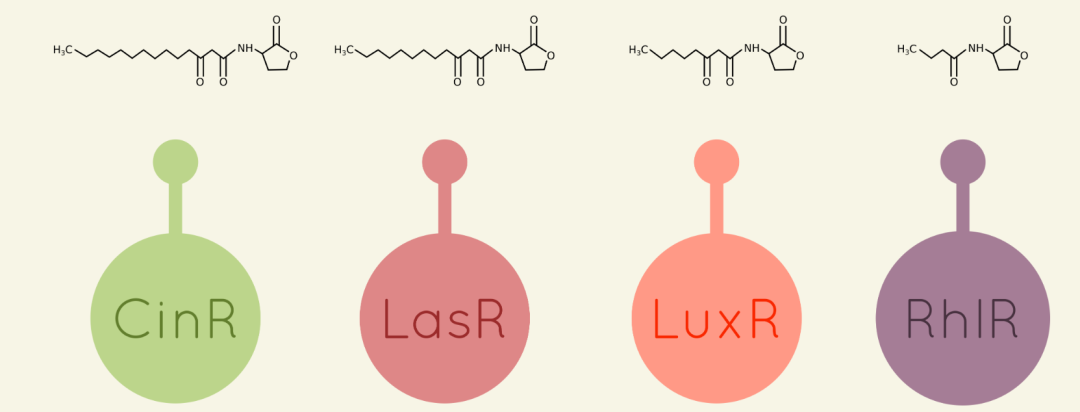
图1 四种群体感应蛋白及相关的AHL
2.比较器模块
比较器模块将群体感应信号连接到细胞内RNA逻辑,以便细菌可以将自己的群体与其他细胞系的群体进行比较。
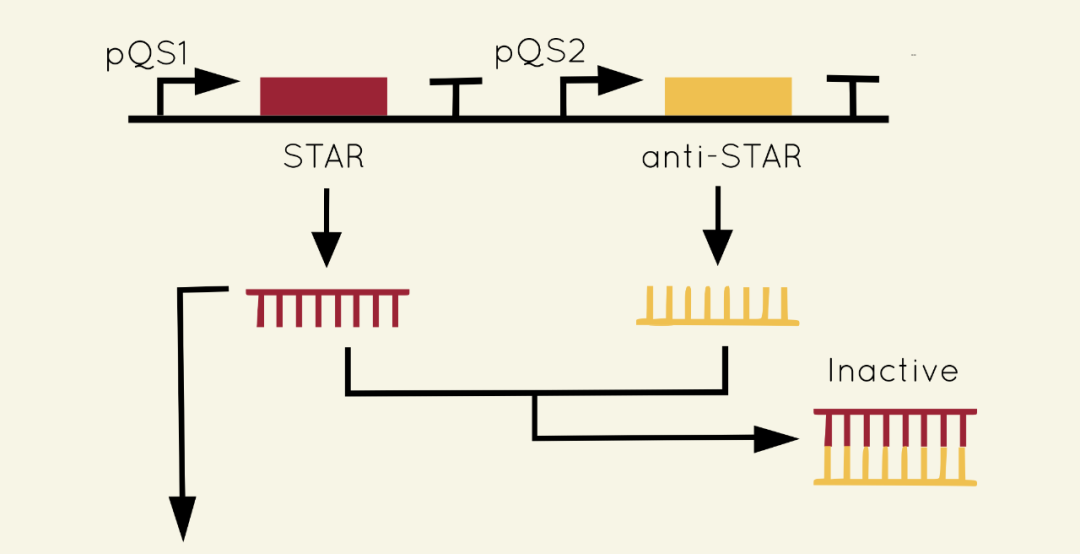
图2 比较器模块SATAR设计原理图
本模块核心设计应用了small transcription activating RNAs(STAR)调控系统。在细胞内,目标基因上游存在发卡结构终止子pAD1结合于上游启动子区域,遏制RNA聚合酶的结合,而当STAR存在时,STAR可与发卡结构终止子pAD1互补配对,解除抑制作用,结果来看,激活了目标基因的转录。
本课题在STAR序列上游放置群体调节启动子pQS1,在Anti-STAR(一段可以与STAR结合配对RNA序列编码基因)上游放置群体调节启动子pQS2,STAR转录量均取决于群体感应分子浓度,成正比例关系。
如果两个细菌群体大小相同的情况下,群体调节因子诱导的STAR和Anti-STAR量相等,相互结合,则没有多余的STAR作用于pAD1,不会激活生长调节蛋白(抑制生长蛋白)的转录,两个细菌群体处于正常生长状态。如果群体数量不相等,群体规模更大的细胞群分泌更多群体调节因子,作用于基因层面会转录生成更多STAR序列,STAR数量>Anti-STAR,STAR可以发挥解除pAD1抑制的作用,从而激活下游基因转录。
3.生长调节模块
前文所提到的利用STAR系统调控的下游目的基因,作用是要可抑制菌群生长。团队选用的生长抑制蛋白基因有两种:Gp2 ,T7噬菌体基因,抑制减缓细胞生长,可逆性与E.coli RNA聚合酶结合;Gp0.4,T7噬菌体基因,有丝分裂末时(母细胞分裂成两子细胞时)与FtsZ环结合,延缓细胞生长。
优点:快速、可逆、高效。
(1)基因片段小,转导快;
(2)Gp2表达后蛋白直接作用于聚合酶,起到抑制作用,无需多级调控,线路反应迅速;
(3)相较于LeuB营养缺陷型细胞难制作,成本高的特点,表达Gp2不需过多流程和成本;
(4)避免抗生素耐药问题;
(5)普通培养基培养即可。
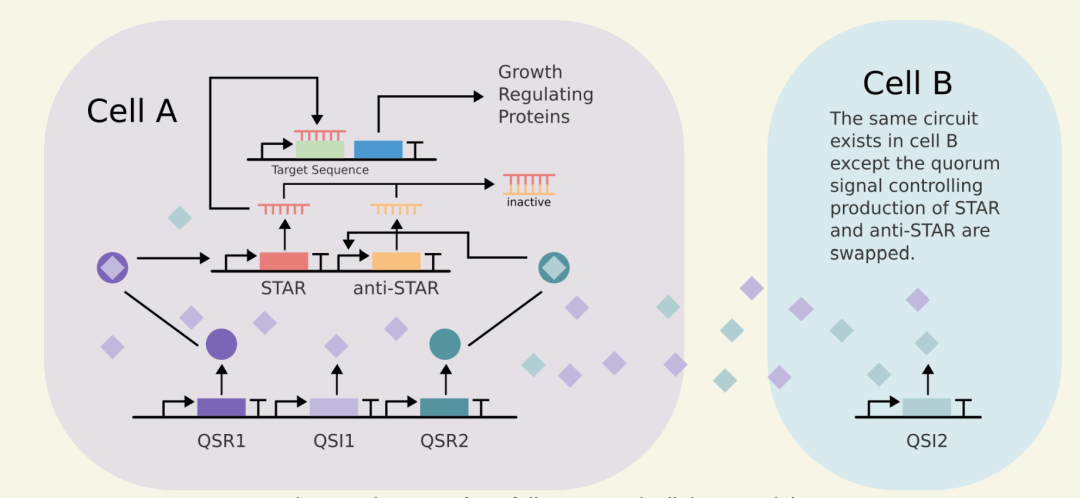
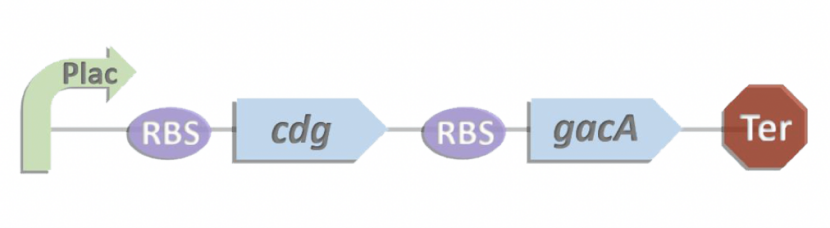
图3 基因线路设计图
4.A.L.I.C.E.
Imperial_College针对共培养体系条件难确定的问题,设计了A.L.I.C.E.,一个可用于指导菌株共培养条件与实验方法的数据库软件。
Part.03课题结果
results
Part 1: 通讯模块
实验1:检查不同群体感应系统间是否会产生串扰
分别将Las, Rhl, Cin, Lux四种群体感应系统彼此之间进行正交性测定(即检查相互之间是否会产生信号干扰)。由于时间原因,该队伍只来得及做了Las和Rhl的正交性测定。
方法:
构建如下图所示的质粒并转入:
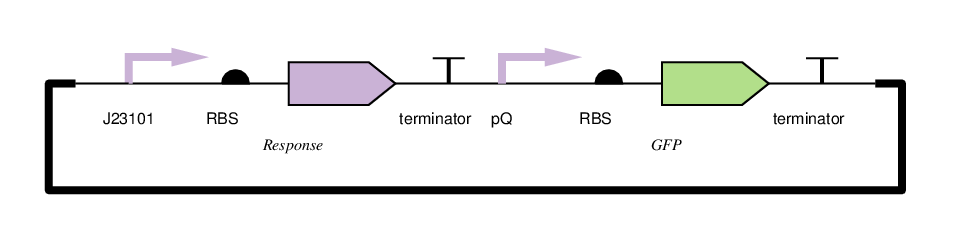
图1 质粒表征结构设计,其中Response 代表群体感应系统对应的转录激活因子,pQ为群体启动子
实验将转入了不同转录激活因子的质粒的细菌培养到指数增长阶段之后转移到96孔板中并在180分钟后监测荧光信号和吸光度值,计算单个细胞的归一化荧光度值。
结果
对于Rhl系统:
其自身的C4 AHL激活其转录激活因子所需的浓度约为100nM-100uM,且检验表明Rhl会与Lux高度串扰,与Las轻微串扰,而与CinR几乎无串扰。
对于Las系统:
其自身的C12 AHL激活其转录激活因子所需的浓度约为100pM-10uM,且检验表明Las会与Cin高度串扰,与Lux和Rhl均几乎无串扰。
综上所述,Rhl与Las正交性较高,能够作为一组正交群体感应系统在实验中发挥作用。
Part 2:比较器模块
实验2:STAR系统的功能验证实验
STAR系统能够结合目标基因上游的pAD1质粒衰减子序列,即本实验中的生长抑制基因。pAD1 质粒衰减子序列能够形成一个RNA发夹结构阻止 RNA 聚合酶转录基因,充当转录终止子。STAR会与该发夹序列结合并干扰其形成,从而允许下游基因的转录。
方法:
为了验证其功能,设计了两个质粒:
1.基本STAR质粒
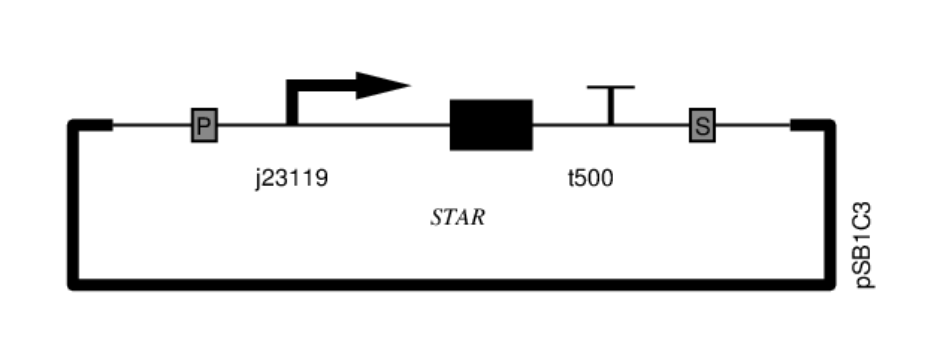
图4 基本STAR质粒示意图
存在该质粒的细胞中有STAR RNA存在,能够解除pAD1的抑制并转录其下游基因。
2.报告质粒
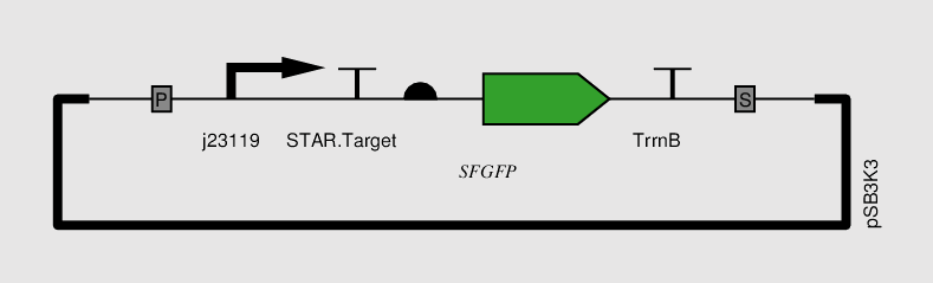
图5 报告质粒示意图
若存在此质粒的细胞中有STAR RNA的存在,则sfGFP的表达限制将被解除,能够发出绿色荧光。
首先,为了确定STAR能够在多大程度上激活 sfGFP 转录,在 200 分钟内连续记录 sfGFP 在同时具有两种质粒的细胞中的荧光,以及在仅具有报告质粒的细胞中的荧光。
之后,为研究STAR系统在不同温度下的功能,分别在30℃和37℃条件下分别将同时具有两种质粒的细胞以及仅具有报告质粒的细胞培养5小时并测定其OD600 = 0.4条件下的荧光值。
结果
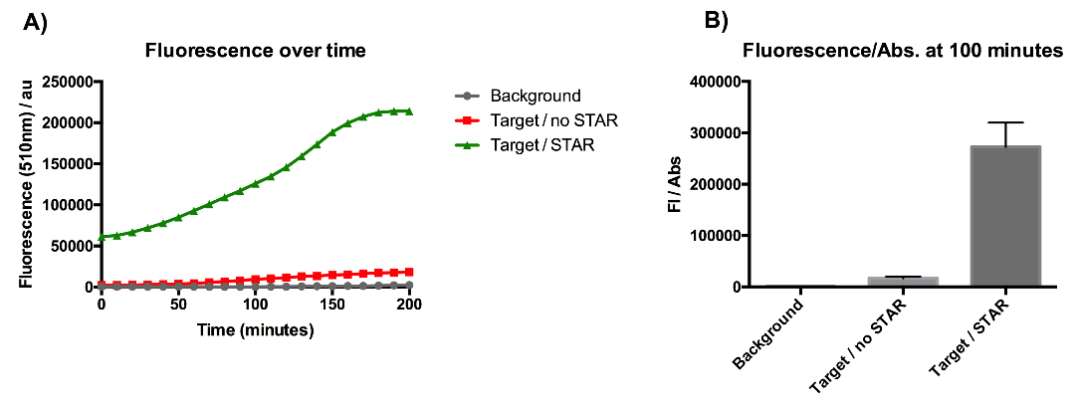
图6 荧光度测定值示意图
A中表示在200分钟内,双质粒组的总荧光度稳定上升;B中表示100分钟时存在STAR的质粒组归一化荧光度远高于单报告质粒组,这表明 STAR 成功地阻止了 pAD1 质粒衰减器干扰 sfGFP 转录。
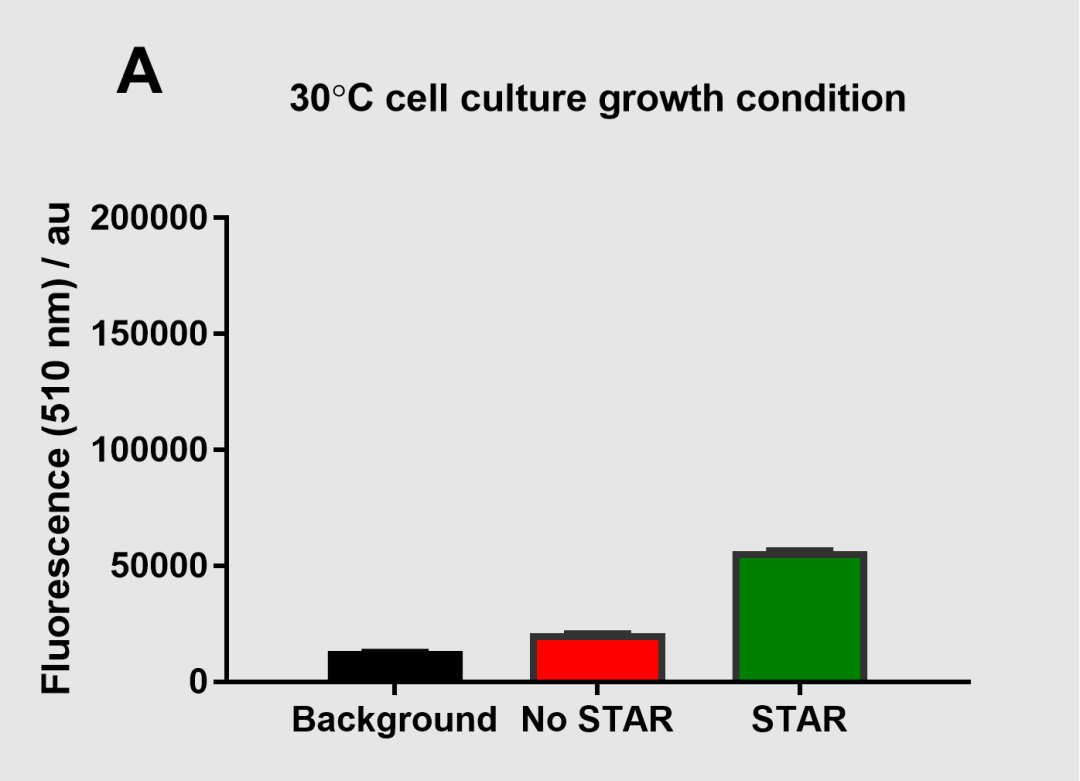
图7 30℃条件下的细胞培养荧光测定
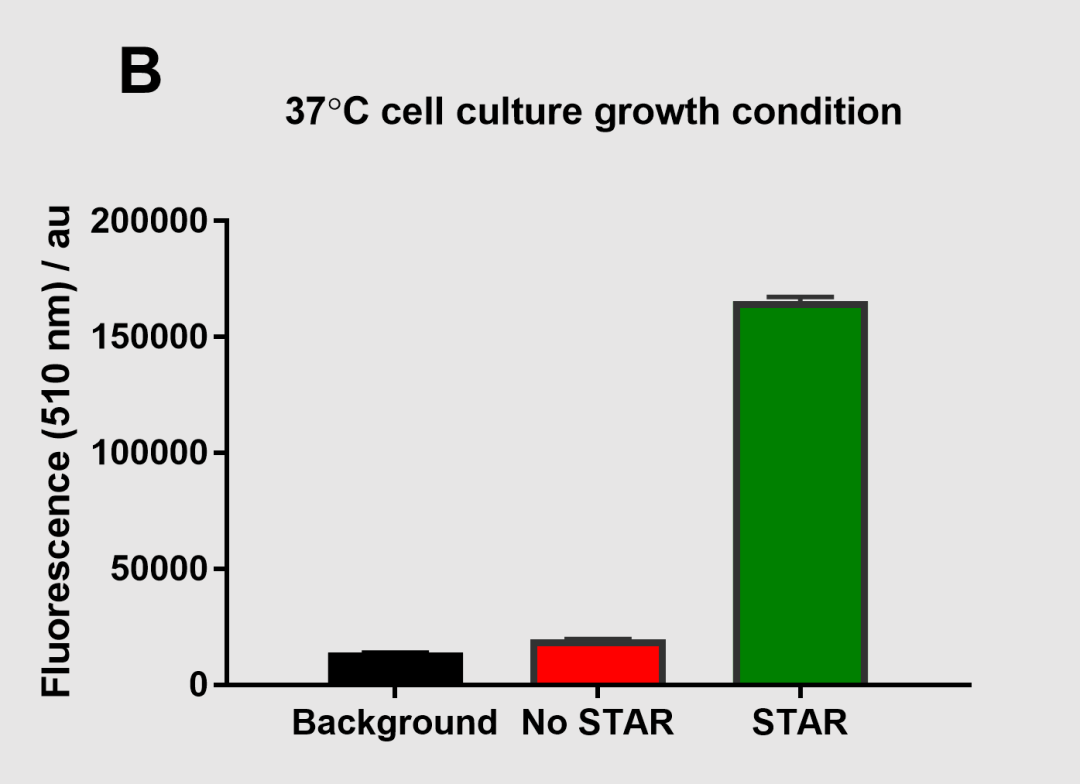
图8 37℃条件下的细胞培养荧光测定
用 STAR 系统可获得的激活 sfGFP 表达要大得多,表明在不同的培养条件下使用STAR系统的效果也不同。
Part 3. 生长调节模块
实验3:生长抑制蛋白Gp2能否有效抑制菌群生长及其可逆性验证
方法:实验将Gp2基因置于阿拉伯糖操纵子下调控表达,在用阿拉伯糖诱导和停止诱导情况下,观察Gp2表达,以及对于菌群生长抑制效果。
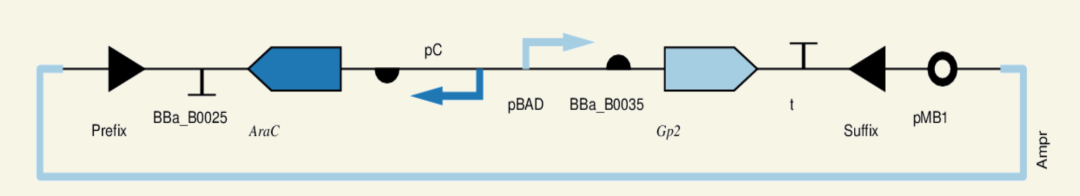
图9 阿拉伯糖诱导Gp2表达质粒构建
知识补充—阿拉伯糖操纵子
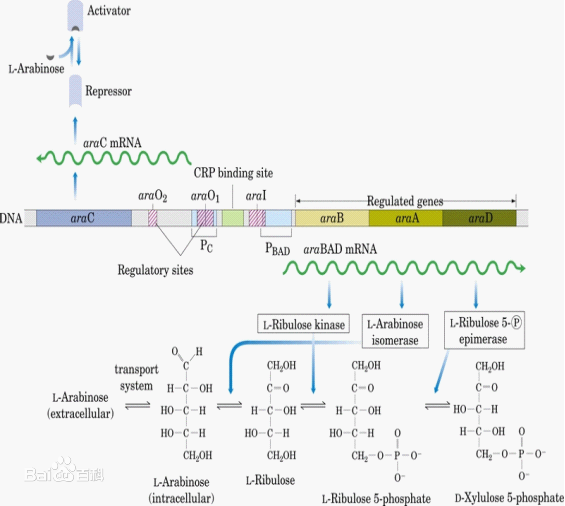
图10 阿拉伯糖操纵子原理示意图(图源百度百科)
当Glu和Ara都存在时,araC本底转录,产生少量的araC蛋白,结合于araO1(-106~-144),使RNA聚合酶不能结合araPC,使araC的转录受到阻遏。
当有Ara存在,而没有Glu时,Ara可作为糖源。此时Ara和少量的araC蛋白结合形成了诱导型的araC蛋白,它作为正调控因子结合于araI,促进了araPBAD的转录,产生了3种酶,促使Ara分解;
当Ara不存在或者用过完了,过量的araC蛋白可以结合则araO1上,阻碍RNA聚合酶在此区域结合,从而关闭了操纵子;或者结合到araI(-40~-78)和araO2上,彼此相互作用形成了环,阻遏了PBAD和PC的启动。
结果
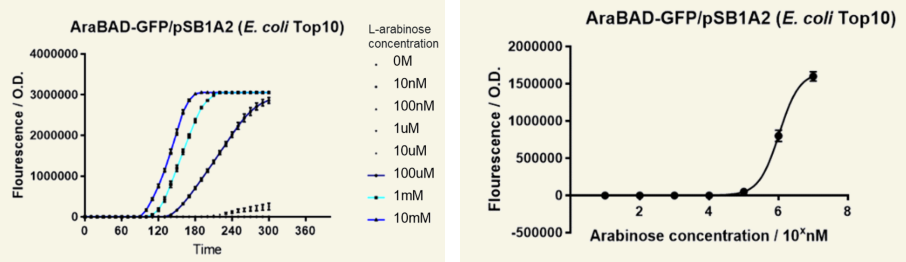
图11 AeaBAD-GFP/pSB1A2荧光强度表达
左图,37℃,不同浓度阿拉伯糖诱导细菌,荧光强度随时间变化,10 mM阿拉伯糖诱导荧光强度最强。右图,荧光强度随阿拉伯糖浓度诱导变化,表明阿拉伯糖浓度与荧光强度呈现正相关。
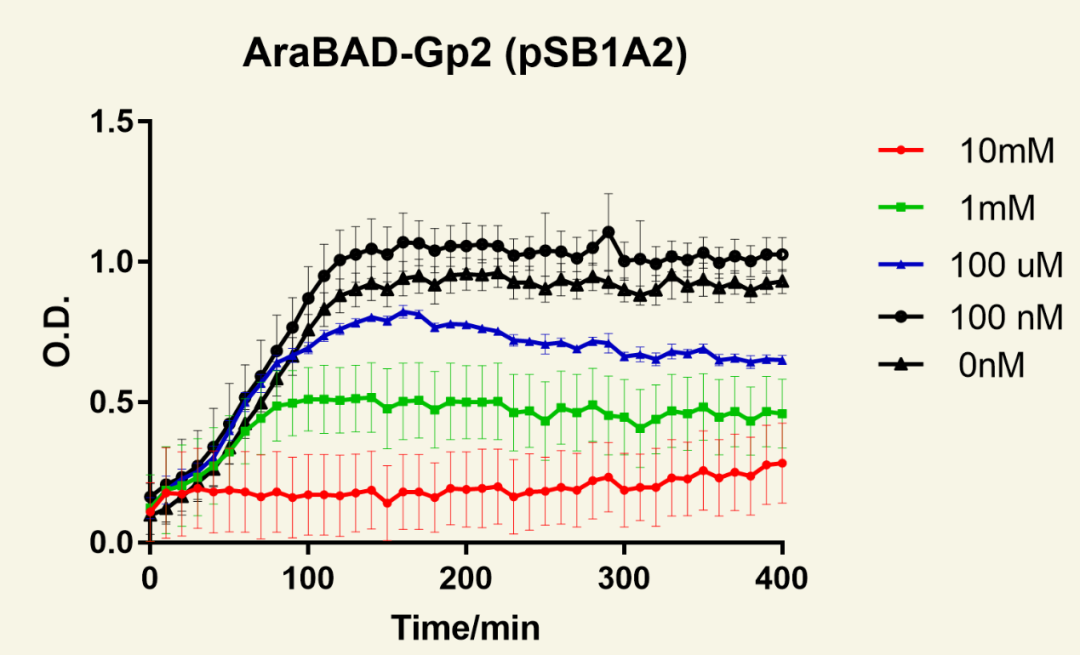
图12 不同浓度阿拉伯糖诱导下菌群随时间生长情况
将Gp2置于ara操纵子下调控,诱导后细菌生长情况。结果可见,10 mM ara诱导后,对菌群生长抑制效果最好。
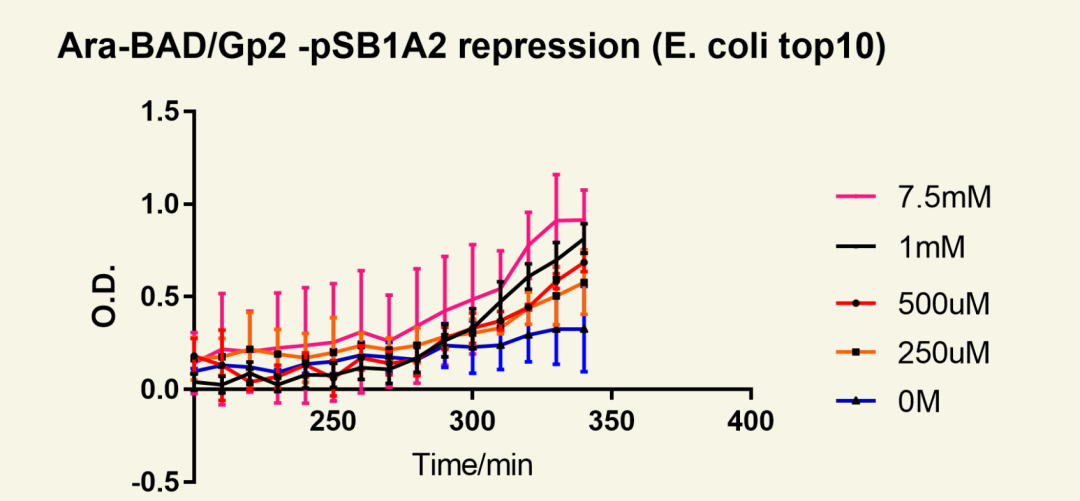
图13 停止阿拉伯糖诱导后菌群随时间生长情况
葡萄糖诱导,以关闭阿拉伯糖操纵子。结果显示,菌群生长曲线呈现明显上升趋势,表明菌群自大约250 min开始菌群生长恢复。
Part.04社会实践human practices
在Imperial College 的项目中,运用了社会技术整合协议(STIR),这意味着考虑项目的伦理、社会、法律或环境维度,并将这些维度纳入项目工作。通常,iGEM基础研究赛道的队伍只会与其他科学家沟通工作;通过STIR这种方式反思实验室的日常决策,反思对现实世界的应用能力,通常能够让社会问题得到更优的解决方案。