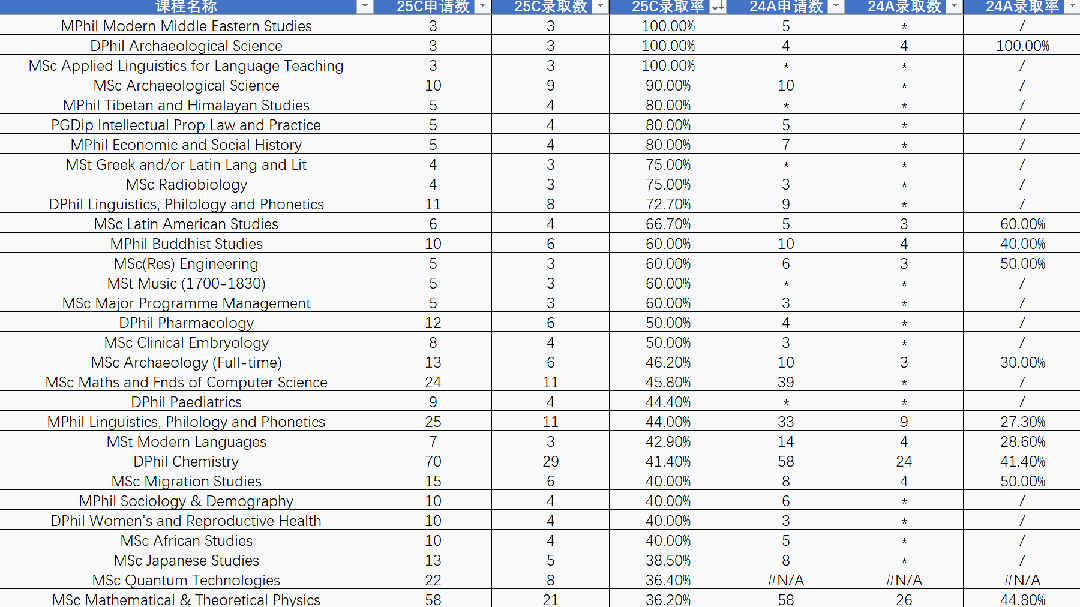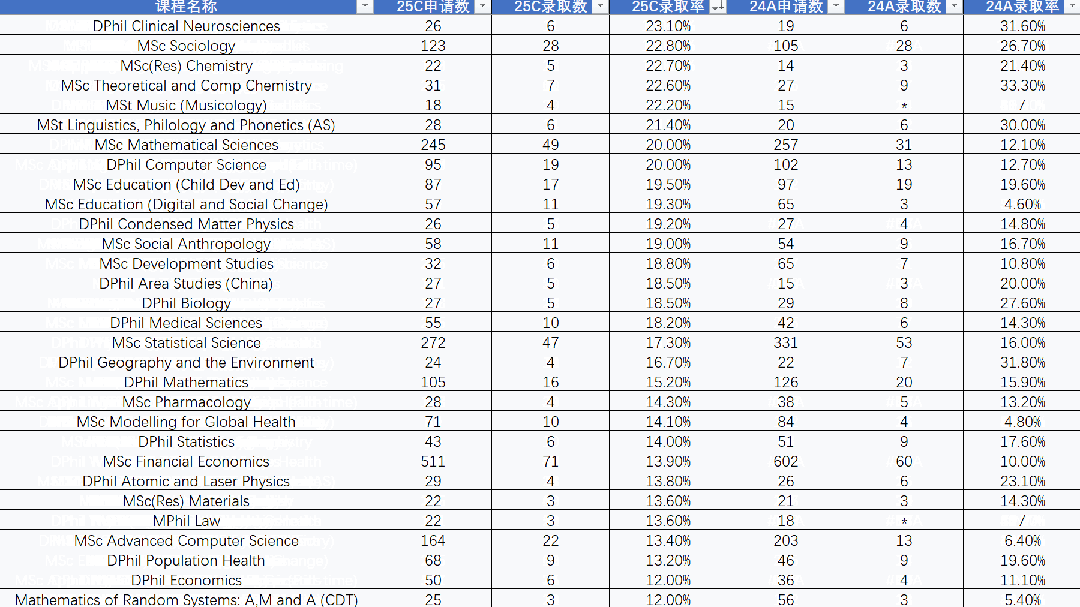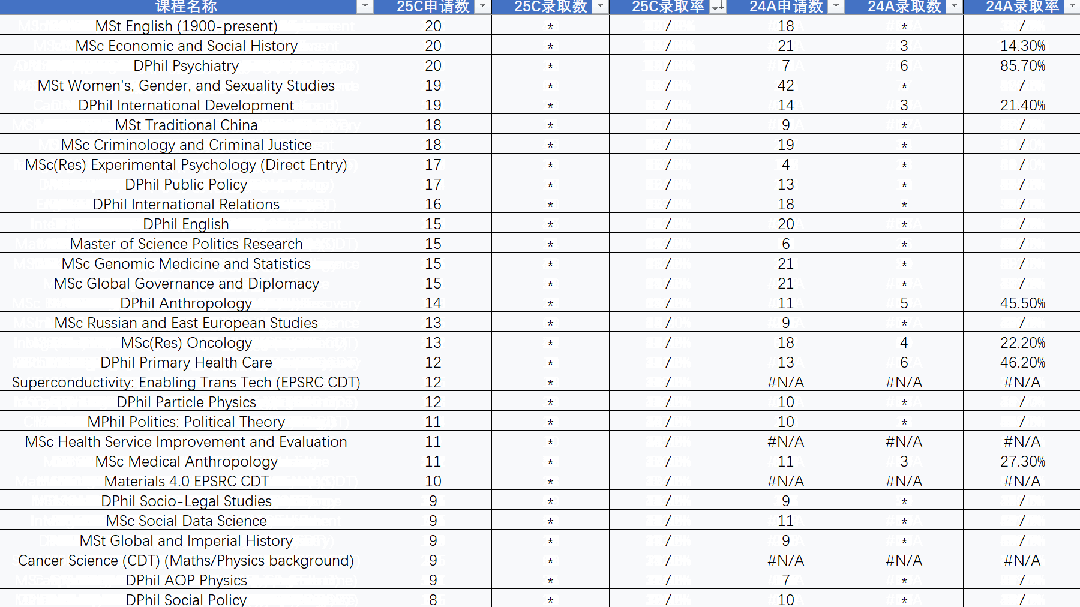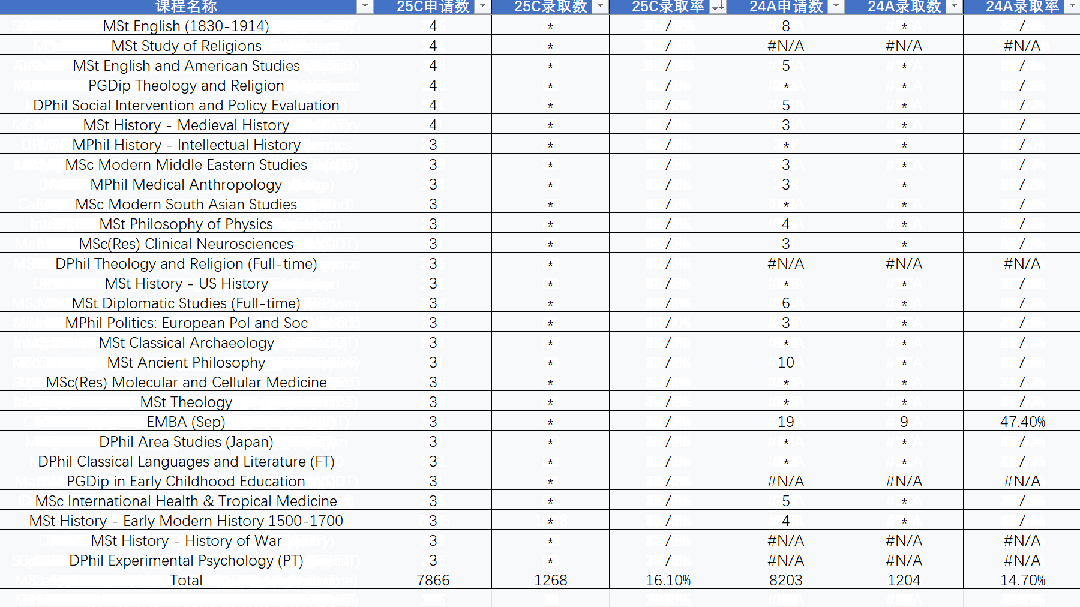
GeoLifeCLEF 2024 @ LifeCLEF & CVPR-FGVC 基于地理位置的 物种存在预测竞赛
竞赛名称:
GeoLifeCLEF 2024@ LifeCLEF & CVPR-FGVC
基于地理位置的物种存在预测竞赛
竞赛类型:计算机视觉
竞赛目的:这一挑战旨在利用各种可能的预测因子来预测给定地点和时间的植物物种:包括卫星图像和时间序列、气候时间序列以及其他栅格化环境数据:地貌、人类活动痕迹、生物气候和土壤变量。
竞赛网址:https://www.kaggle.com/competitions/geolifeclef-2024/overview
赛题背景
在空间和时间上预测植物物种组成及其变化,对于许多与生物多样性管理和保护相关的场景、改进物种识别和清单工具以及教育目的都是有用的。
这一挑战旨在利用各种可能的预测因子来预测给定地点和时间的植物物种:包括卫星图像和时间序列、气候时间序列以及其他栅格化环境数据:地貌、人类活动痕迹、生物气候和土壤变量。
为此,我们提供了一个大规模的训练集,其中包括约500万个欧洲地区的植物出现数据(单标签、仅存在数据),以及约5,000个地块的验证集和包含20,000个地块的测试集,其中包含所有当前物种(多标签、存在-缺失数据)。
该挑战的难点包括从单一正标签进行多标签学习、强烈的类别不平衡、多模态学习和大规模学习。
赛题时间
2023年12月:LifeCLEF挑战的注册开始(免费)
2024年2月28日:发布训练和测试数据
2024年5月24日:竞赛截止日期。
2024年6月7日:提交工作说明论文的截止日期[CEUR-WS会议论文]。
2024年6月21日:接受通知 - 工作说明论文[CEUR-WS会议论文]。
2024年7月8日:工作说明论文的最终版本截止日期。
2024年9月9日至12日:CLEF 2024在法国格勒诺布尔举行。
动机 MOtivation
预测特定位置的植物物种对许多生物多样性管理和保护场景都有帮助。
首先,它可以构建物种组成和相关生物多样性指标(如物种多样性、濒危物种和入侵物种)的高分辨率地图。在科学生态学中,这个问题被称为物种分布建模。
此外,它还可以显著提高物种识别工具的准确性 - 如Pl@ntNet - 通过减少在给定地点可观察到的候选物种列表。
更普遍地说,它可以通过开发基于位置的推荐服务(例如在手机上),鼓励公民科学家观察者的参与,并加速物种观察的注释和验证,以产生大规模、高质量的数据集来促进生物多样性清单。
最后,这可以用于教育目的,通过具有任务或情境化教育路径等功能的生物多样性探索应用程序。
竞赛评估方法
这项竞赛的评估指标是在由物种存在-缺失(PA)样本组成的测试集上计算的样本平均F1分数(在Kaggle上称为F-Score Beta(Micro))。在机器学习术语中,这是一个多标签分类任务。F1分数是预测和实际出现在特定位置和时间的物种集之间重叠的平均度量。
每个测试PA样本i与一组地面真实标签Yi相关联,即与给定列patchID和dayOfYear的组合相关联的植物物种的集合(请参阅数据选项卡以获取有关物种观察数据结构的详细信息)。
对于每个样本,提交将提供一个标签列表,即预测出现的物种集合。

提交格式
对于测试集中的每个id,您必须预测在给定位置出现的一组物种。文件应包含标题,并具有以下格式:
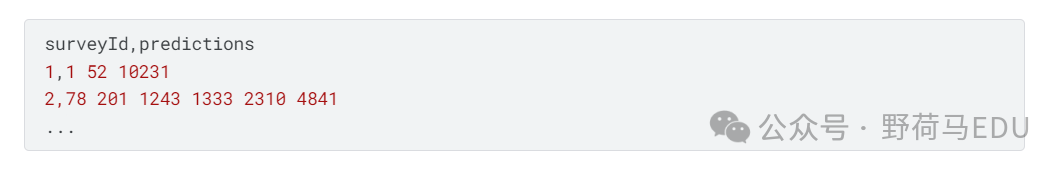
提交格式是一个CSV文件,对于每个样本(行),包含两列:
- surveyId列包含整数,对应于测试样本的id,对应于patchID和dayOfYear列值的唯一组合。
- predictions列包含以空格分隔的预测物种标识符列表(在训练/验证数据集中的列spId)
对于每个样本(行),预测的物种标识符必须按从左到右的值递增的顺序排列。没有测试样本是空的,测试集只包含来自训练或验证集的物种。
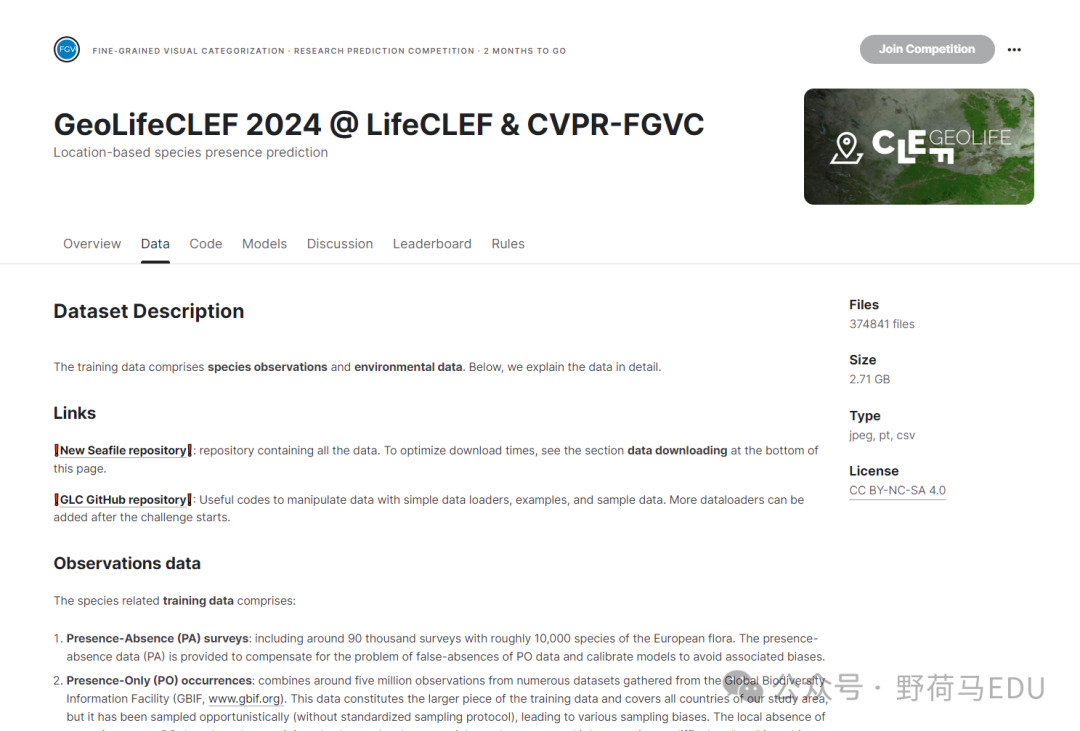
数据集描述
训练数据包括物种观察和环境数据。以下详细解释了数据。
链接
- ❗新的Seafile存储库❗:包含所有数据的存储库。为了优化下载时间,请参见本页面底部的数据下载部分。
- ❗GLC GitHub存储库❗:用于操作数据的有用代码,包括简单的数据加载器、示例和样本数据。挑战开始后可以添加更多数据加载器。
观察数据
与物种相关的训练数据包括:
-
存在-缺失(PA)调查:包括大约9万次调查,涉及大约1万种欧洲植物。存在-缺失数据(PA)用于补偿PO数据的假缺失问题,并校准模型以避免相关偏差。
-
仅存在(PO)观察:结合了来自全球生物多样性信息设施(GBIF,www.gbif.org)的约500万条观察数据,这些数据来自各种数据集。这些数据构成了训练数据的较大部分,并覆盖了研究区域的所有国家,但是它是机会性采样的(没有标准化的采样协议),导致了各种采样偏差。在PO数据中物种的局部缺失并不意味着它真的不存在。观察者可能没有报告它,因为在这个时间点很难“看到”它,将其识别为非监测目标,或者只是不够吸引人。
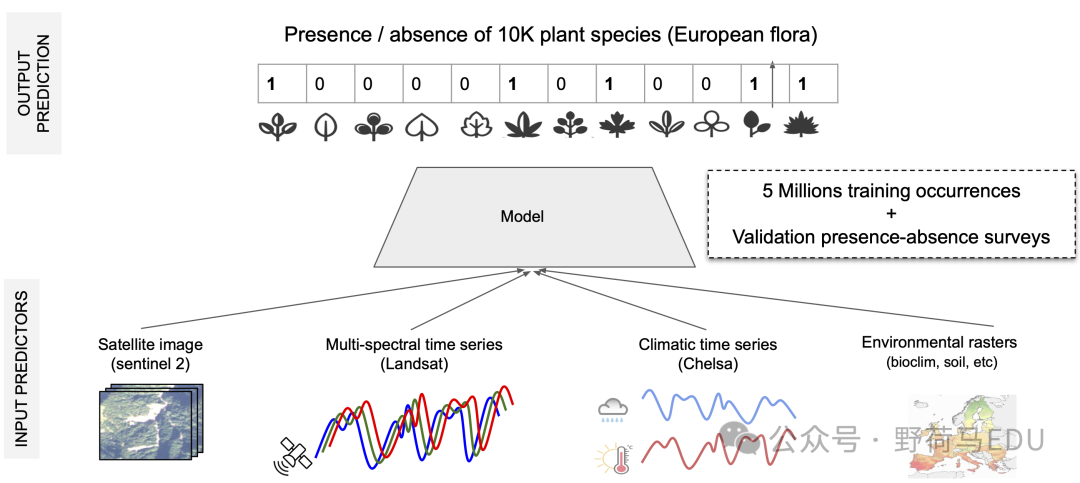
环境数据
除物种数据外,我们还提供了空间化的地理和环境数据作为额外的输入变量(参见图1)。更具体地说,对于每个物种观察位置,我们提供:
- 卫星图像块:3波段(RGB)和1波段(NIR)的128x128图像,分辨率为10m。
- 卫星时间序列:六个卫星波段(R、G、B、NIR、SWIR1和SWIR2)的长达20年的值。
- 环境栅格各种气候、土壤、土地利用和人类足迹变量的欧洲范围。我们提供标量值、时间序列和原始栅格,您可以从中提取本地2D图像。
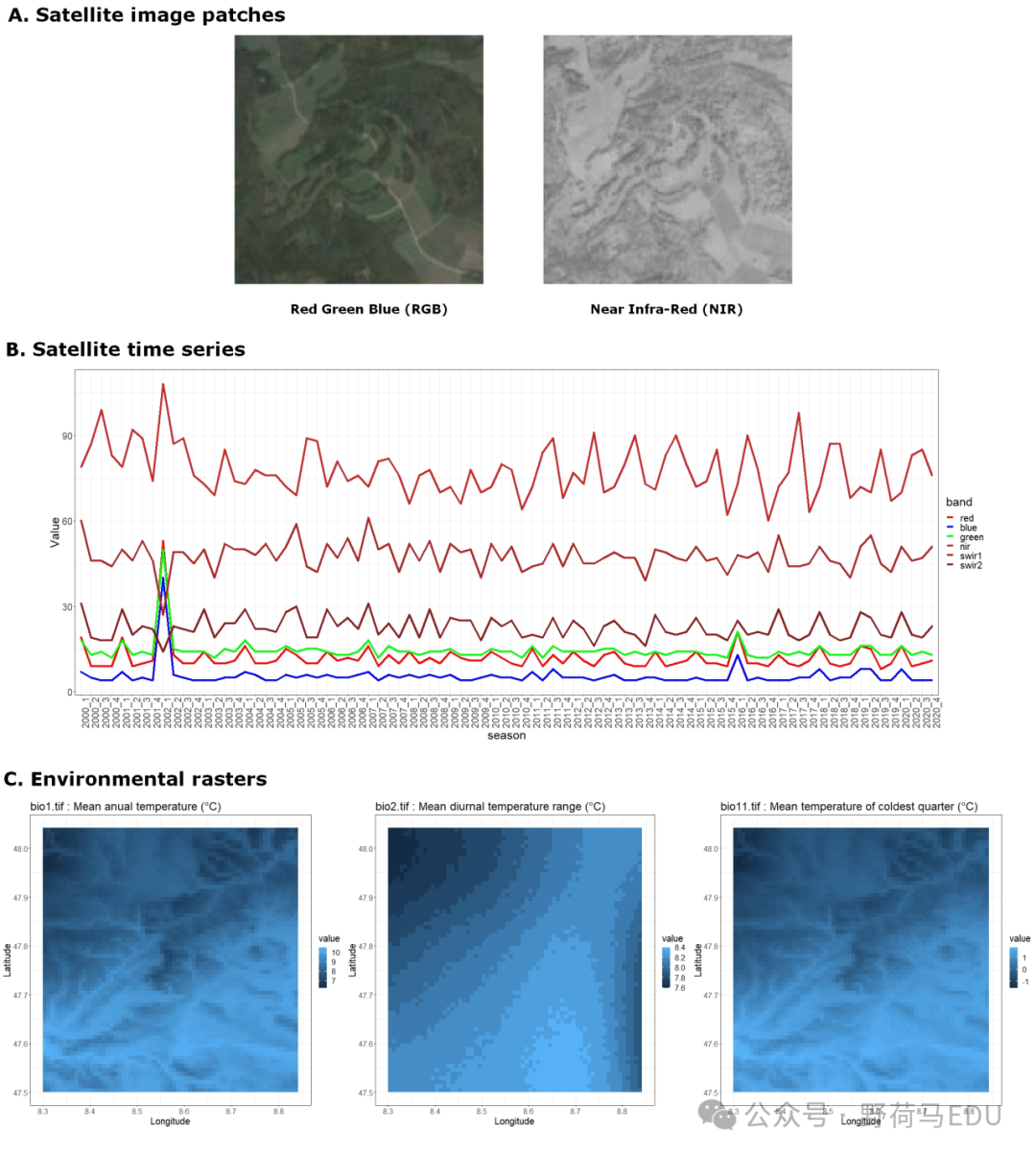
图. 展示了2021年在瑞士北部(经度=8.5744;纬度=47.7704)收集的一个事件(glcID=4859165)的环境数据。A. 在观测周围2021年采样的1280x1280米卫星图像块。B. 自1999-2000年冬季以来,六个卫星波段在该点位置的季度时间序列。C. 从提供的环境栅格中提取的三个生物气候图像(约65x65公里)。
卫星图像块:
1280mx1280m的RGB和NIR图像块(四个波段),以观测地理位置为中心,同年采集。这些图像块被压缩在两个zip文件中(patchs_rgb.zip,patchs_nir.zip),可在/SatelliteImages/文件夹中访问。
- 格式:128x128的JPEG图像,一个用于RGB数据的彩色JPEG文件,一个用于近红外的灰度JPEG文件。
- 分辨率:每像素10米
- 来源:由Ecodatacube平台预处理的Sentinel2遥感数据
- 访问:首先,必须下载并解压提供的zip文件。每个JPEG文件对应于一个唯一的观测位置(通过“surveyId”)。要加载所选观测的RGB或NIR图像块,请从任何事件CSV中获取“surveyId”,并按照以下规则加载它 --> '…/CD/AB/XXXXABCD.jpeg'。例如,surveyId为3018575的图像位置是"./75/85/3018575.jpeg"。对于所有“surveyId”小于四位数的情况,您可以使用类似的规则。对于“surveyId”为1的情况是"./1/1.jpeg"。
卫星时间序列:
每个观测都与自1999年冬季以来每个季节的卫星中位点值的时间序列相关联,涵盖了六个卫星波段(R、G、B、NIR、SWIR1和SWIR2)。这些数据携带了过去20年季节性植被变化、潜在极端自然事件(火灾)或土地利用变化的高分辨率本地特征。
- 格式1:六个CSV文件,每个波段一个。每列代表从2000年冬季到2020年秋季的84个季节。
- 格式2:TimeSeries-Cubes - 上述CSV文件汇总为3D张量,轴为波段、季度和年份。
- 分辨率:原始卫星数据每像素30米
- 来源:由Ecodatacube平台预处理的Landsat遥感数据
- 访问:/SatelliteTimeSeries/
月度气候栅格:
从2000年1月到2019年12月,每月计算的四个气候变量(平均、最低和最高温度以及总降水量),产生了覆盖欧洲的960个低分辨率栅格。
- 格式1:CSV文件,每个栅格一个,通过“surveyId”引用。
- 格式2:TimeSeries-Cubes - 上述CSV文件汇总为3D张量,轴为栅格类型、年份和月份。
- 分辨率:约1公里
- 来源:Chelsa
- 访问:
/EnvironmentalRasters/Climate/Climatic_Monthly_2000-2019
环境栅格:
对于每个观测,我们提供了额外的环境数据,例如GeoTIFF栅格和已从栅格中提取的标量值。我们提供CSV文件,每个波段栅格类型一个,即气候、海拔、人类足迹、土地覆盖和SoilGrids。
生物气候栅格:覆盖欧洲的19个低分辨率栅格;在物种分布建模中常用。以经度/纬度坐标(WGS84)提供。
- 格式:带有压缩的GeoTIFF文件和带有提取值的CSV文件。
- 分辨率:30 arcsec(约1公里)
- 来源:CHELSA
- 访问:
/EnvironmentalRasters/Climate/BioClimatic_Average_1981-2010
土壤栅格:
覆盖欧洲的九个土壤学低分辨率栅格。提供的变量描述了从5到15厘米深度的土壤特性,并决定了植物物种的分布。有关所提供变量的定义,请查看definition.txt文件(例如,pH、黏土、有机碳和氮含量等)。
- 格式:带有压缩的GeoTIFF文件和带有提取值的CSV文件。
- 分辨率:约1公里
- 来源:Soilgrids
- 访问:/EnvironmentalRasters/Soilgrids
- 海拔:覆盖欧洲的高分辨率栅格。
- 格式:带有压缩的GeoTIFF文件,Int16数字存储(13.2GB)和带有提取值的CSV文件。
- 分辨率:1 arc second(约30米)
- 来源:ASTER Global Digital Elevation Model V3
- 访问:/EnvironmentalRasters/Elevation
土地覆盖:
覆盖欧洲的中分辨率多波段土地覆盖栅格。每个波段描述了土地覆盖类别的预测或在各种分类下的置信度。我们推荐使用IGBP(17个类别)或LCCS(43个类别)图层,这些图层经常用于物种分布建模。
- 格式:带有压缩的GeoTIFF文件和带有提取值的CSV文件。
- 分辨率:约500米
- 来源:MODIS Terra+Aqua 500米
- 访问:/EnvironmentalRasters/LandCover/
人类足迹:
提供了几个描述人类足迹的低分辨率栅格,涵盖了由人类存在和活动引起的环境七大压力(例如夜间光照水平、人口密度)的两个时期,分别是90年代初(约1993年)和2000年代末(约2009年)。我们提供了两个总结栅格,结合了所有人类压力,以及每个压力的两个详细栅格,这些栅格避免了原始数据的任意降级。
- 格式:带有压缩的GeoTIFF文件和带有提取值的CSV文件。
- 分辨率:约1千米
- 来源:Venter等人,2016年
- 访问:
/EnvironmentalRasters/HumanFootprint/文件夹中包含了一个详细介绍此数据的readme文件和两个子文件夹:summarized/ 包含了两个总结栅格(分别为1993年和2009年),以及 detailed/ 包含了2*7个单一压力栅格。