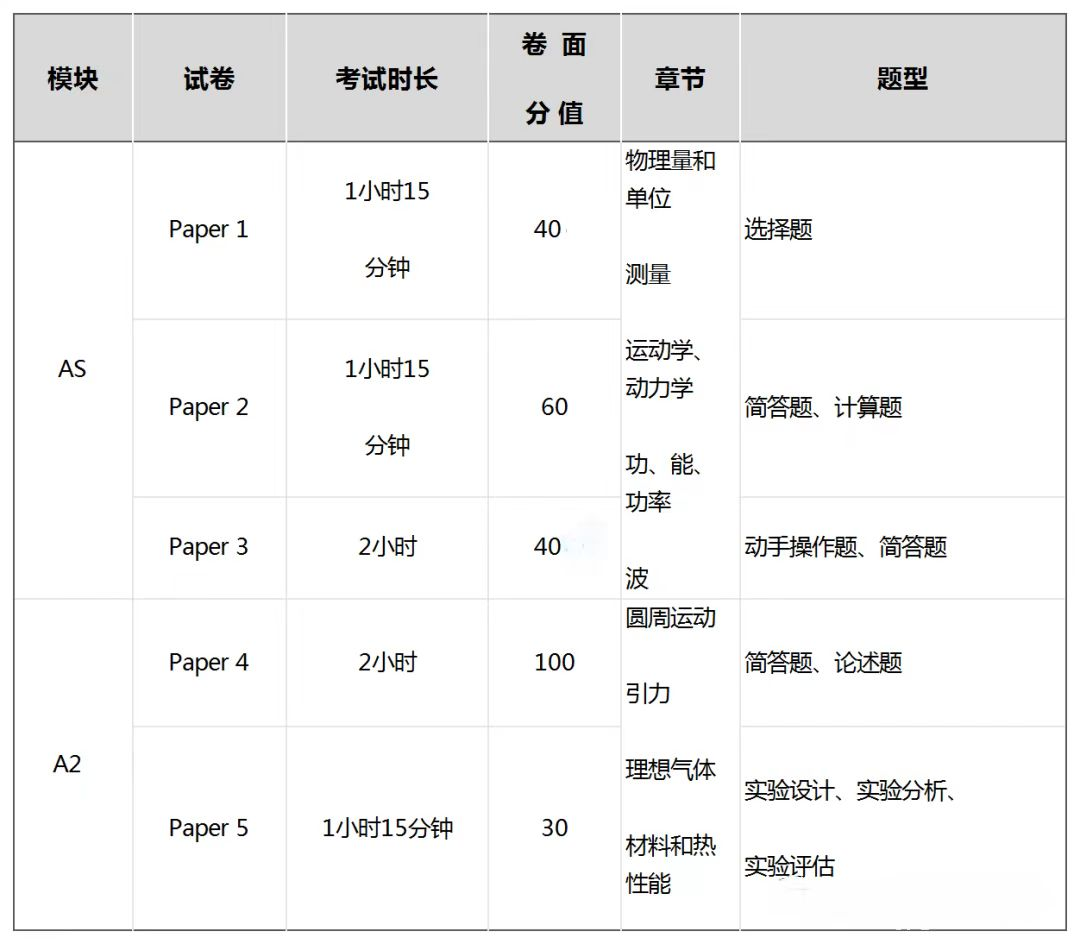
文献分享
Information storage across a microbial community using universal RNA barcoding
Prashant B. Kalvapalle, Lauren Gambill, August Staubus, Matthew J. Dysart, Kiara Reyes Gamas, Jonathan J. Silberg, Li Chieh Lu, Lauren B. Stadler, James Chappell
基因转移(如接合、转导和转化)是微生物进化、生态功能塑造及抗生素抗性传播的核心机制。它影响细菌的生态位扩展、噬菌体抗性演化,并推动合成生物学中工程菌株的开发。然而传统研究方法在复杂微生物群落中存在诸多局限性。近期,一项创新研究开发了一种基于催化RNA(cat-RNA)的自主记录系统,为基因转移监测带来了新的突破,让我们一起来看看这项研究的精彩内容!
No.1
研究背景:
传统基因转移研究方法(如荧光蛋白或抗生素抗性基因标记)依赖宿主细胞的生长和翻译,在复杂微生物群落中灵敏度不足,且无法实时监测跨物种的基因转移动态。此外,基于DNA的标记技术(如转座子插入)需依赖宿主基因组整合和宏基因组测序,存在操作复杂性和灵敏度限制。
现有挑战
01灵敏度不足
传统方法难以检测低丰度或非可培养微生物的基因转移事件。
02依赖翻译
荧光报告基因需宿主表达系统,限制了在多样化微生物中的应用。
03操作复杂性
化学或酶标记技术需人工干预,无法自主记录自然环境中的基因转移。
针对这些问题,本研究开发了一种基于催化RNA(cat-RNA)的自主记录系统,通过靶向高度保守的16S rRNA区域,将合成条形码写入rRNA。该方法无需翻译,直接通过测序分析即可识别参与基因转移的微生物类群,显著提高了灵敏度和适用范围。
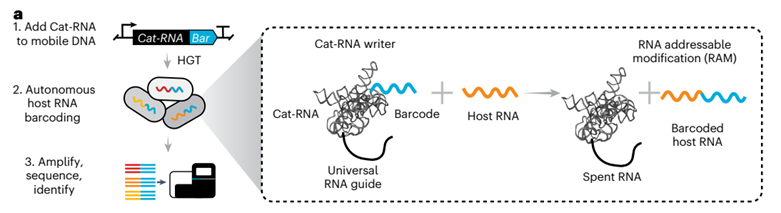
No.2
研究思路与结果:
实验首先设计了一个由催化RNA核心、可编程RNA引导序列和非编码RNA(ncRNA)条形码组成的荧光报告系统,通过将核酶引导序列靶向绿色荧光蛋白(gFP)基因的特定区域(gfp-F1),当核酶催化RNA剪接反应后,可将ncRNA条形码(gfp-F2)连接到目标RNA上,形成完整的gFP转录本,从而通过荧光信号验证系统活性。为优化引导序列长度,研究团队构建了不同长度(0-600 nt)的核酶变体,在E. coli中发现50 nt引导序列时荧光强度最高(达阳性对照的30%),且对细胞生长无显著影响(补充图3)。
进一步在Vibrio natriegens(海洋环境)、Pseudomonas putida(土壤)、Shewanella oneidensis(淡水)等不同门类微生物中测试,结果显示所有物种均能产生显著高于阴性对照的荧光信号(Welch检验,P≤0.045),表明核酶系统无需物种特异性优化即可跨微生物运行。
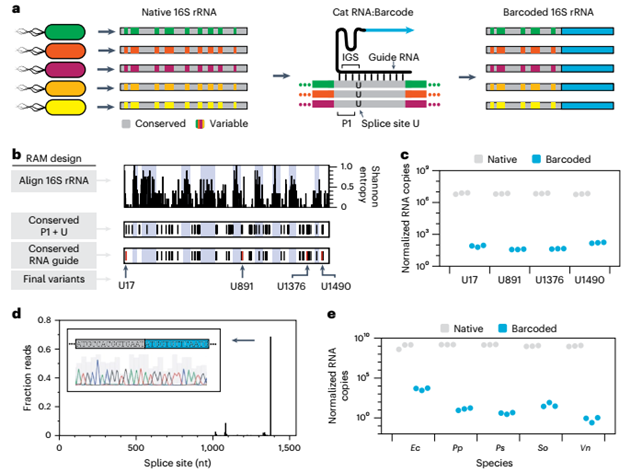
接着,研究团队通过多序列比对分析E. coli、P. stutzeri等5种微生物的16S rRNA序列,筛选出4个高度保守的尿嘧啶位点(U17、U891、U1376、U1490),并设计对应核酶的内部引导序列(IGS),使其与目标位点上游5 nt形成P1茎结构。通过RT-qPCR定量,发现靶向U1376的核酶在E. coli中效率最高,相当于每个细胞约2条修饰分子。
跨物种测试显示,尽管核酶在P. putida中的活性仅为E. coli的20%,但在所有测试菌株中均检测到显著信号,证明保守位点设计策略的有效性。此外,通过反向PCR与高通量测序验证剪接特异性,发现95%的修饰发生在目标U1376位点,仅少数脱靶事件涉及相似P1结构的邻近尿嘧啶。稳定性实验表明修饰rRNA半衰期为3-8分钟,与典型mRNA相当,表明其适合记录动态基因转移事件。
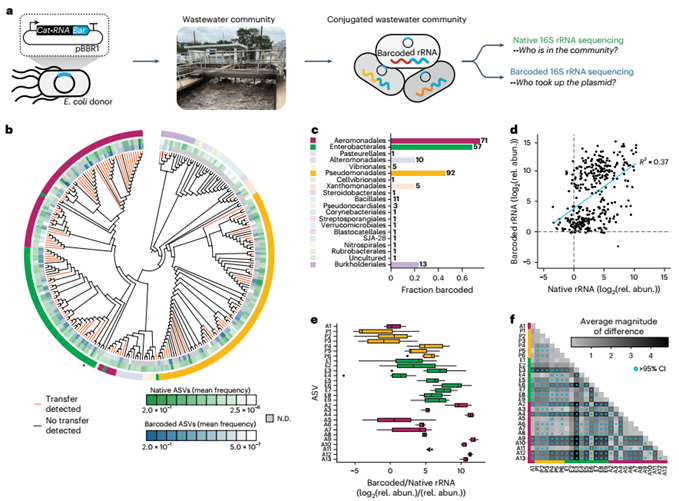
接下来,研究者使用携带CymR抑制系统的E. coli MFDpir作为供体,通过滤膜接合法与休斯顿某污水处理厂的活性污泥群落共培养24小时。对总RNA进行反向转录后,采用针对16S rRNA V6-V8区的通用引物和靶向条形码的特异性引物分别扩增,通过Illumina测序解析群落组成。
结果显示,在检测到的279个扩增子序列变异(ASV)中,140个显示条形码信号,涵盖20个细菌目,其中20个ASV仅在修饰rRNA中检出,表明这些低丰度类群可能具有高接合效率。通过比较ASV丰度与条形码信号的相关性,发现两者呈弱正相关,但部分高丰度ASV信号强度显著低于预期,表明质粒稳定性或核酶表达效率的物种差异。
更深入的分析显示,同一目内ASV的条形码/总rRNA比值差异可达43,000倍(如肠杆菌目内),通过Bootstrap检验确认63%的ASV对存在显著差异(P<0.05),揭示基因转移效率受接合机制、宿主限制修饰系统等多因素调控。
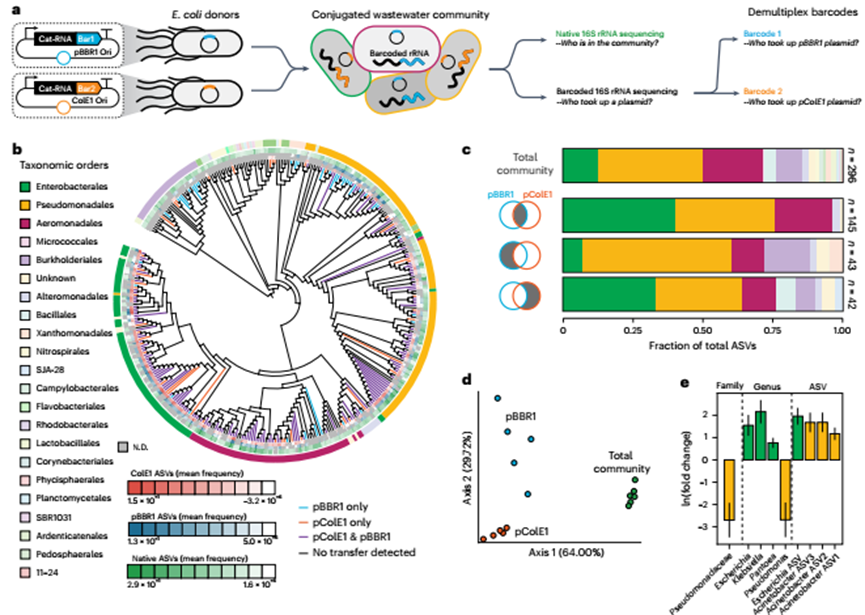
最后,研究团队通过多路复用核酶系统,比较不同复制起点质粒的宿主范围。研究设计了携带pBBR1和ColE1复制起点的正交核酶质粒,分别靶向16S rRNA的U1376位点并连接不同条形码序列。
将两种供体菌混合后与废水群落接合,测序分析显示pBBR1质粒标记188个ASV,ColE1标记187个ASV,其中145个重叠,但两者宿主分布存在显著差异:pBBR1在肠杆菌目中占主导,而ColE1在假单胞菌目中信号强度仅为pBBR1的15%,主坐标分析(PCoA)进一步证实两种质粒的宿主群落结构分离,与已知ColE1在γ-变形菌中偏好性分布一致。
该实验首次实现单一样本内平行追踪多质粒动态,揭示复制起点对宿主范围的调控作用,为设计环境适应性载体提供关键数据。
No.3
研究意义与展望:
研究通过开发基于合成催化RNA(cat-RNA)的通用标记技术,在微生物群落水平实现了基因转移事件的实时记录与宿主范围分析,具有重要科学意义和应用价值
传统依赖荧光蛋白或抗生素抗性基因的报告系统受限于宿主翻译能力,且需细胞培养扩增,无法灵敏检测复杂群落中的低丰度基因转移事件。本研究通过靶向微生物高度保守的16S rRNA,利用cat-RNA直接在RNA分子上写入可测序的条形码信息,无需翻译即可记录基因转移。
实验证明该方法可检测到传统荧光报告系统(如mScarlet)遗漏的20%的宿主分类单元,灵敏度显著提升,且能捕获低丰度微生物的基因转移行为。
同时,该技术可通过设计靶向特定RNA序列的cat-RNA,选择性标记不同微生物类群或遗传元件,实时追踪工程菌株在自然环境中的基因泄漏风险。例如,结合CRISPR系统可开发自毁机制,限制合成基因扩散。此外,基于RNA的信息存储具有可逆性,避免了DNA整合对宿主基因组的永久性改变,为动态调控微生物群落功能提供了可控手段,推动了合成生物学安全性与环境工程的应用。