XMU-CHINA
1. 课题基本信息
课题名称:
Undergrad Gold Medal — BreFast: A Fast and Convenient Screening Kit for Breast Cancer Early Detection
主要内容:
团队结合 CRISPR/Cas13a 技术和侧向层析流动试纸创建了一种快速便捷的筛查试剂盒——BreFast,用于乳腺癌的早期检测。
Wiki 网址:
https://2023.igem.wiki/cjuh-jlu-china/
关键词:
CRISPR/Cas13a技术、侧向层析流动试纸、乳腺癌检测
2. 课题背景
乳腺癌是女性发病率最高、死亡率最高的癌症。其中,乳腺癌的大多数死亡与晚期发现有关,导致患者错过最佳治疗期。目前,乳腺癌的治疗主要包括手术、放疗、化疗和靶向治疗,而这些治疗方法通常对早期乳腺癌患者非常有效,对于晚期患者并不总能达到预期的效果;统计显示,大约30%的乳腺癌患者因癌症转移而死亡。
在这种情况下,乳腺癌筛查旨在发现癌细胞以早期干预癌症进展,使患者的生存期明显延长。通过筛查早期发现乳腺癌可以显著改善患者预后,因为它增加了保留患者乳房的可能性,加速康复过程,降低并发症的概率,并有助于预防与疾病相关的心理健康问题。尤为关键的是,乳腺癌发现得越早,患者接受化疗的概率就越低,患者不必面对从心脏毒性到骨髓毒性的化疗副作用。
但传统的乳腺癌诊断方法如侵入性组织活检常常伴随着患者的疼痛。因此,为了实现早期乳腺癌检测以及鼓励更多人接受定期乳腺癌检测,团队设计了 BreFast——一种快速便捷的乳腺癌筛查试剂盒。对比许多传统基于 qRT-PCR的 miRNA 检测技术(其耗时、昂贵且需要可靠的实验技能),BreFast 利用 CRISPR/Cas13a 系统的灵敏度和侧流条带的便利性,从而使得乳腺癌筛查变得容易。
3. 课题原理
1. miRNA
miRNA 是一种含有 21~23 个核苷酸的小型单链非编码 RNA 分子,存在于细胞内和血液中,能够与 mRNA 中的互补序列结合,调节基因表达和细胞功能。
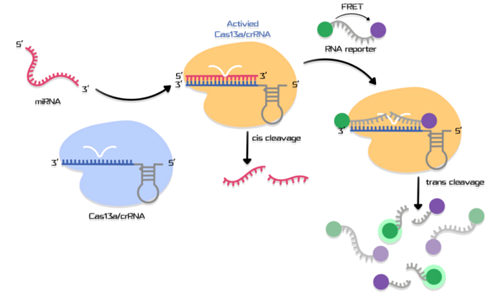
图1. miRNA 与 mRNA 结合调节基因表达和细胞功能
通过生物信息学分析确定了乳腺癌的一个合适生物标志物,miR-21-5p。
2. CRISPR/Cas13a 系统
CRISPR 是原核生物的一种用来抵抗噬菌体的免疫系统:原核生物含有来自噬菌体的 DNA 序列片段,这些 DNA 片段转录出的 RNA用作 gRNA 来检测具有相同序列的噬菌体 DNA。这些 gRNA 与 Cas 蛋白结合后,可与含有互补序列的噬菌体 DNA 形成 RNA-DNA 双链,从而激活 Cas 蛋白的核酸酶活性,切割噬菌体 DNA,进而抵抗噬菌体。
如何构建一个针对 miR-21-5p 的 CRISPR/Cas13a 切割系统:
Cas13a 识别 crRNA 的环状区域,并形成 Cas13a/crRNA 复合物;随后,crRNA 与含有互补序列的目标 RNA 形成双链 RNA,通过二者的相互作用激活 Cas13a 的核酸酶活性。激活后的 Cas13a/crRNA 复合物不仅切割目标 RNA,还会非特异性地切割其他任意 RNA 序列(无论是否与 crRNA 互补)。

图2. CRISPR/Cas13a 系统工作原理
3. 侧向层析流动试纸
侧向层析流动试纸是一种基于抗原与抗体特异性结合的快速检测方法,具有操作简便、经济的特点,且结果通常在 30 分钟内显示。
当样品加至样品垫上时,样品沿着条带流动,经过探头垫到达测试区,最后到达吸收垫。如果报告基因未被切割,存储在探头垫上的金标记链霉亲和素将结合到报告基因的生物素侧,并随之沿着条带迁移,随后,位于 T 线的抗 FAM 抗体捕获报告基因的 FAM 侧,导致胶体金(用红色表示)积累;反之,如果报告基因被切割,生物素侧与 FAM 侧发生分离,因此 T 线的抗 FAM 抗体无法捕获生物素以及结合到生物素的胶体金。
因此,根据 T 线胶体金的存在,可以很容易地知道报告基因是否被切割。此外,质量控制线(线 C)上的抗体可以捕获金标记的小鼠抗体,从而证明样品已顺利通过整个条带。
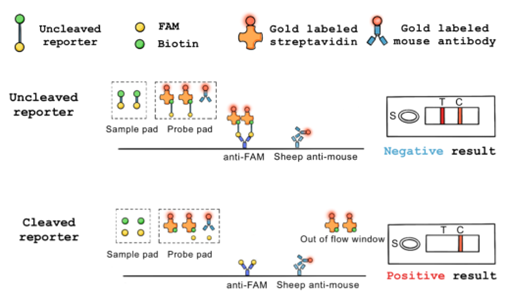
图3. 侧向层析流动试纸的原理
将 CRISPR/Cas13a 系统与侧向层析流动试纸结合,区分 miR-21-5p 表达水平高或低的样本,以实现对乳腺癌的筛查。
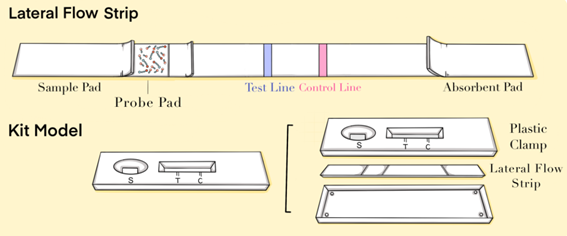
图4. 侧向层析流动试纸示意图
4. BreFast
BreFast 采用工程化的 crRNA 特异性靶向 miR-21-5p;当 crRNA 与目标 miRNA 结合时,Cas13a 的核酸酶活性被激活,导致报告 RNA 被切割;切割的报告 RNA 可以通过侧向层析流动试纸检测出其相对丰度,反映样本中 miR-21-5p 的表达水平相关,从而检测诊断乳腺癌。
实际操作:将样本与 Cas13a/crRNA 复合物混合,如果样本中含有 miR-21-5p,则导致报告 RNA 的切割;随后,将 Cas13a 反应混合物滴加到侧向层析流动试纸上;最后,条上的两条带表示阴性,而一条带表示 miR-21-5p 阳性。阳性结果意味着乳腺癌的高风险,建议去医院进行进一步诊断,以确认他们是否患有乳腺癌。
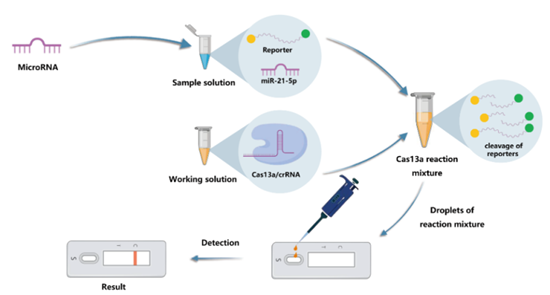
图5. BreFast 的应用图
4. 项目设计
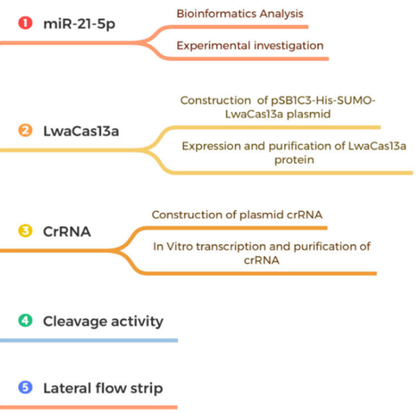
图6. 该课题设计的内容
(一)miR-21-5p
1. 生物信息学分析
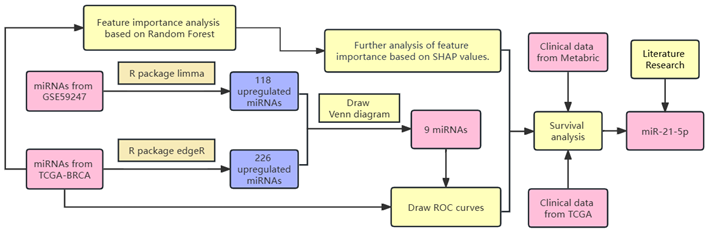
图7. 生物信息学分析方法流程
(1)差异表达分析
为了确定在乳腺癌患者中表达显著变化的 miRNA,分析 TCGA 数据库中的 TCGA-BRCA 数据集和 GEO 数据库中的 GSE59247 数据集,并选择了|logFC|>1 和 PValue<0.05 阈值的 miRNA。
从 GSE59247 数据集中选出 118 个上调和 98 个下调的 miRNA,从 TCGA-BRCA 数据集中选出 226 个上调和 107 个下调的 miRNA,再根据二者之间相交部分选出 9 个 miRNA。
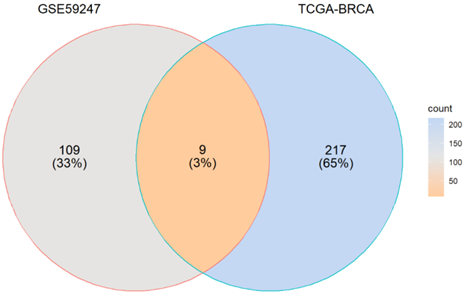
图8. 两个数据集中上调的 miRNA 数量的韦恩图
(2)ROC曲线
为进一步筛选检测的目标 miRNA ,绘制了上述 9 个 miRNA 的 ROC 曲线。由于 GSE59247 数据集中的样本量相对较小,使用 TCGA-BRCA 数据集来生成 ROC 曲线。
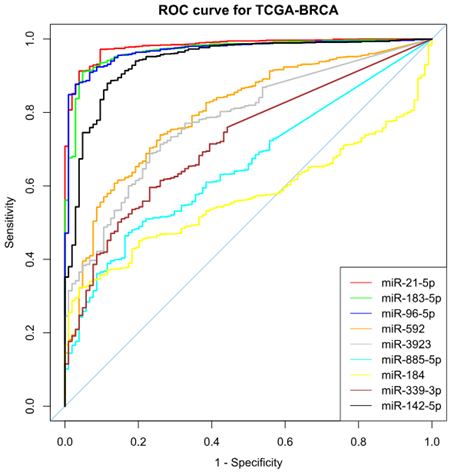
图9. 从 TCGA-BRCA 数据生成的 ROC 曲线
以 AUC>0.9 作为筛选标准,选出 miR-21-5p、miR-183-5p 和 miR-96-5p 进行后续分析。
(3)特征重要性分析
特征重要性分析是一种机器学习方法,用于分析某个特征对特定特征(标签)的重要性。
将每个 miRNA 视为一个特征,并根据样本是否来自癌症患者或正常人将其标记为阳性或阴性,从而将选择最佳癌症相关 miRNA 的问题转化为机器学习特征重要性问题。数据集按照 8:2 的比例分为训练数据和测试数据;使用网格搜索和五折交叉验证来寻找最佳参数(在五折交叉验证中实现了 0.98924 的准确率)。
使用随机森林模型执行二分类任务,在测试数据集上的准确率达到1,其中特征重要性是基于基尼指数计算的。
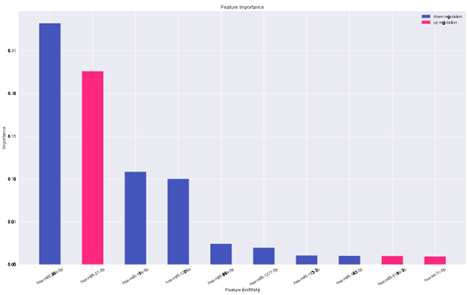
图10. 随机森林分析
考虑到不同 miRNA 之间可能存在强烈的相互作用,采用了基于合作博弈论的 SHAP(SHapley Additive explanations)分析方法,对随机森林模型中得到的特征重要性进行进一步分析。
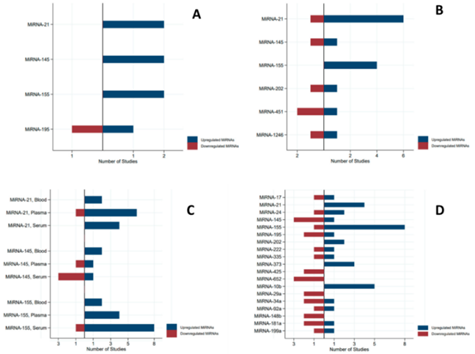
图11. SHAP 分析
(4)生存分析
验证 miR-21-5p 是否真的能影响乳腺癌的进展,使用 Kaplan-Meier 曲线对 miR-21-5p 进行生存分析,使用 Metabric 和 TCGA 的数据绘制 miR-21-5p 的生存曲线。
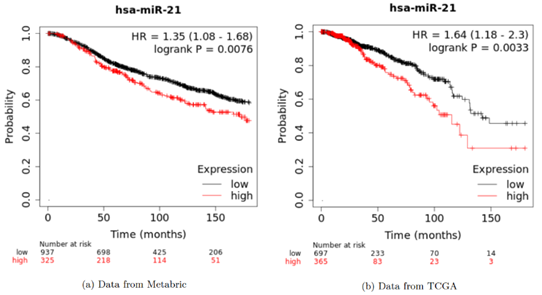
图12. miR-21-5p 的生存曲线
结果显示,miR-21-5p 高表达乳腺癌患者的生存时间短于低表达患者,且 miR-21-5p 与乳腺癌患者的生存时间之间存在统计学上的显著相关性。
(5)文献研究
除了生物信息学分析外,团队阅读文献以确认 miR-21-5p 是否作为乳腺癌相关生物标志物的最佳 miRNA 。
miR-21、miR-155、miR-10b 被多份研究支持,与乳腺癌高度相关,且值得注意的是,miR-21 的主要形式是 miR-21-5p。

图13. miRNA 表达方向(乳腺癌病例与对照 miRNA 浓度)的锥形图
(A)全血;(B)血浆;(C)所有标本;(D)血清。
结果表明,miR-21-5p 是检测乳腺癌的最佳相关 miRNA。
(1)miR-21-5p 在乳腺癌细胞中的表达
进行 qRT-PCR 检测,以确定 miR-21-5p 在乳腺上皮细胞系 MCF10A 和乳腺癌细胞系 MCF7 中的相对表达量。
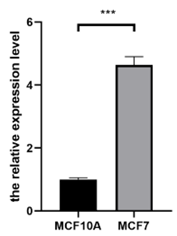
图14. MCF10A 和 MCF7 细胞中 miR-21-5p 的表达
(2)miR-21-5p 对乳腺癌细胞迁移和侵袭的影响
1)miR-21-5p 对乳腺癌细胞侵袭能力的影响
首先进行 Transwell 实验。
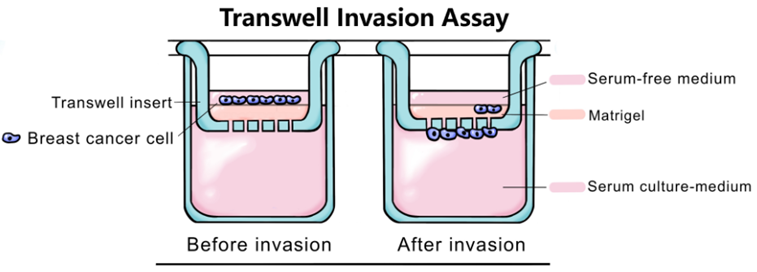
图15. Transwell 侵袭实验示意图
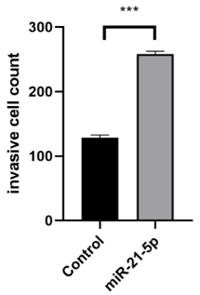
图16. Transwell 侵袭实验结果
与转染对照 RNA 的细胞相比,转染 miR-21-5p 的 MDA-MB-231 细胞的侵袭能力显著增强,表明 miR-21-5p 增加了 MDA-MB-231 细胞的侵袭性。
2) miR-21-5p 对乳腺癌细胞迁移的影响
伤口愈合实验:实验组MDA-MB-231 细胞被转染 miR-21-5p 、对照组 RNA,并在 0 小时、24 小时和 48 小时记录细胞迁移情况。
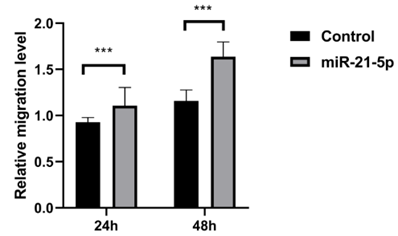
图17. 相对迁移水平(p<0.001)实验结果
与转染对照 RNA 的 MDA-MB-231 细胞相比,转染 miR-21-5p 组的伤口面积显著减小,表明 miR-21-5p 增强了 MDA-MB-231 细胞的迁移能力。
综上结果表明:miR-21-5p 表达增加对乳腺癌细胞具有显著影响。
5. 实验设计
(二)LwaCas13a
1.构建 pSB1C3-His-SUMO-LwaCas13a 质粒
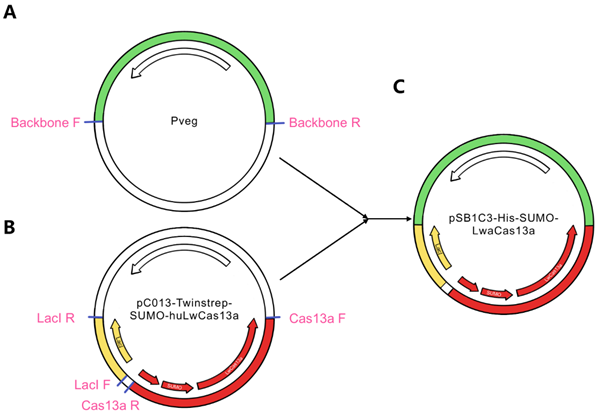
图18. pSB1C3-His-SUMO-LwaCas13a 克隆策略
2.表达与纯化 LwaCas13a 蛋白
实验方法:
SDS-PAGE 凝胶电泳;
Western blot;
使用 His-Tag 蛋白纯化试剂盒和 IDA-Ni 磁性琼脂糖珠进行纯化,并使用 SUMO 蛋白酶 ULP1 消化 SUMO-LwaCas13a。
(三)crRNA
1.crRNA的设计
crRNA 由两部分组成,一个包含环状区域的重复部分和一个包含线性引导序列以识别目标 RNA 的检测部分。研究表明,crRNA 引导序列的长度会影响其活性;当 crRNA 引导序列的长度小于 20 nt 时,其活性显著降低。故设计了四个与 miR-21-5p 互补的 crRNA 引导序列,其长度范围为 20 nt 至 22 nt。
图19. crRNA 的二级结构
(A)crRNA1、(B)crRNA2、(C)crRNA3、(D)全长 crRNA
2. 构建 pSB1C3-crRNA 质粒
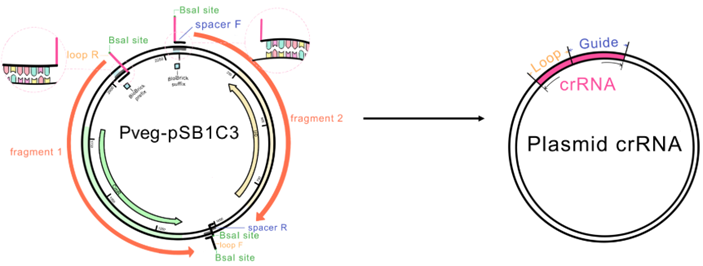
图20. crRNA 序列的 Pveg 质粒克隆策略示意图
3.体外 crRNA 转录与纯化
4.检测不同crRNA 和 Cas13a 蛋白组装的 CRISPR 反应系统
荧光反应具有高灵敏度和定量特性,因此选择通过荧光检测来分析 CRISPR/Cas13a 系统的切割活性。
使用一种基于荧光共振能量转移(FRET)原理的报告 RNA,一端带有 FAM 基团,另一端带有黑洞猝灭剂组(BHQ1),中间通过6个核苷酸连接;激活的 CRISPR/Cas13a 系统可以在连接处切割报告 RNA,并释放 FAM 基团从猝灭剂组中分离出来;释放的 FAM 基团发出的荧光可以被检测到。
将设计的 crRNA 和 LwaCas13a 构建形成功能性复合物,使得该复合物的切割活性可以被 miR-21-5p 激活,从而切割报告 RNA 以释放 FAM 基团进行荧光检测。
(1)测量不同crRNA效果
实验使用四个 crRNA。
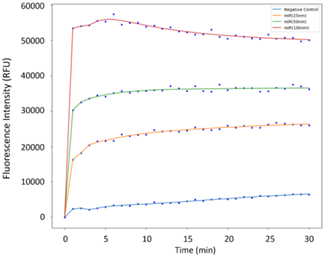
图21. 不同 crRNAs 在不同时间测量的荧光强度
结果表明,由全长 crRNA 和 Cas13a 蛋白组装的 CRISPR 反应系统具有最强的切割活性,其次是 crRNA3,而 crRNA2 和 crRNA3 活性最弱。
(2)miR-21-5p 浓度的影响
crRNA、LwaCas13a 和报告 RNA 的浓度分别固定在 20 nM、100 nM 和 100 nM,而 miR-21-5p 的浓度从 0 nM 逐渐增加到 100 nM。

图22. 不同时间不同浓度 miRNA 测量的荧光强度
结果表明,随着 miRNA 浓度的增加,荧光强度也随之增加。
(3)报告 RNA 浓度的影响
设置 0 nM、100 nM、125 nM、250 nM 和 500 nM 的一系列报告基因浓度进行实验。
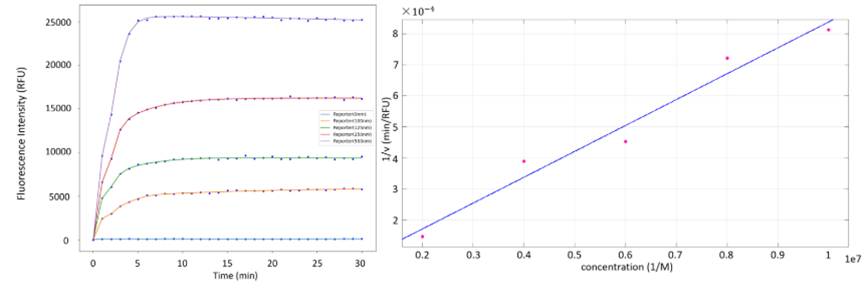
图23.
(A)不同时间不同浓度报告 RNA 测量的荧光强度
(B)林德曼-伯克拟合曲线。
实验结果发现与 Michaelis-Menten 方程一致,这与我们的预期相符。
(四)侧向层析流动试纸
在 miR-21-5p 未表达的情况下:CRISPR/Cas13a 系统未激活,RNA 报告基因未被切割。此时,未被切割的 RNA 报告基因与链霉亲和素标记的胶体金结合,进而在 T 线位置被 FAM 抗体捕获;同时,金标记的鼠抗体在 C 线位置被羊抗鼠抗体捕获。因此,横向流动条带显示 C 线和 T 线,表明阴性结果。
在 miR-21-5p 表达低的情况下:CRISPR/Cas13a 系统未完全激活,报告基因未完全切割。未切割的 RNA 报告基因与链霉亲和素标记的胶体金仍可被 FAM 抗体在 T 线位置捕获;而切割的 RNA 报告基因则不能再与链霉亲和素标记的胶体金和 FAM 抗体连接。因此,横向流动条显示 C 线以及微弱的 T 线,表明阴性结果。
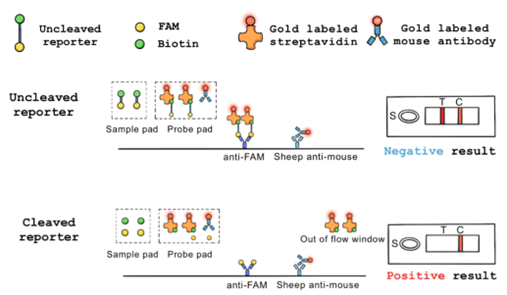
图24/3. 侧向层析流动试纸的原理图
进行实验:改变 miR-21-5p 的浓度进行检测。

图25. 成功区分 miRNA 的浓度
结果表明,该试纸能有效检测 miR-21-5p。
6. 硬件开发
设计制造了侧向层析流动试纸的外壳,使其便于携带和使用。
图26/4. 侧向层析流动试纸示意图
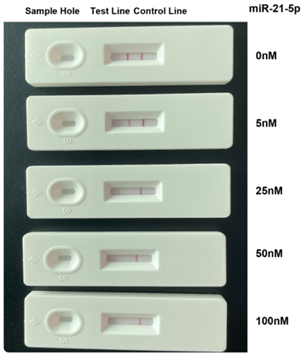
图27. 最终成体图
7. 个人总结
该课题利用CRISPR技术应用在检测领域(进行乳腺癌早期筛查),设计包括利用生物信息学去筛选鉴定出一个在乳腺癌患者中表达变化显著的 miRNA,从交叉数据库分析 → 机器学习特征筛选 → 实验功能验证 → 临床文献佐证。
可以看出往年已有利用CRISPR技术去检测的例子,那么,就算检测是蛋白质或是其他,又或是原理不同,我们课题设计应用偏向检测方向不太适合。因为一是新颖度不够,二是我们课题是关于细菌体内转录或转录后调控,如果检测肯定是体外检测效率更高更便捷,将检测物与细菌体内链接起来优势不大,所以最终排除掉CRISPR应用做检测方向。